はじめに †
Rを用いてネットワーク構造を可視化したので,そのメモです.
準備 †
igraphパッケージを使います.
install.packages("igraph") library("igraph")
データの読み込み †
ネットワークは隣接行列で表現します. そこで,隣接行列をCSV形式で作成し,Rに読み込みます. ここでは,1行目と1列目には頂点の名前が入っているものとします.
data <- read.csv("data.csv", header=T, row.names=1)
これを隣接行列に変換します. ここでは無向グラフ("undirected")とします.
g <- graph.adjacency(data,mode="undirected")
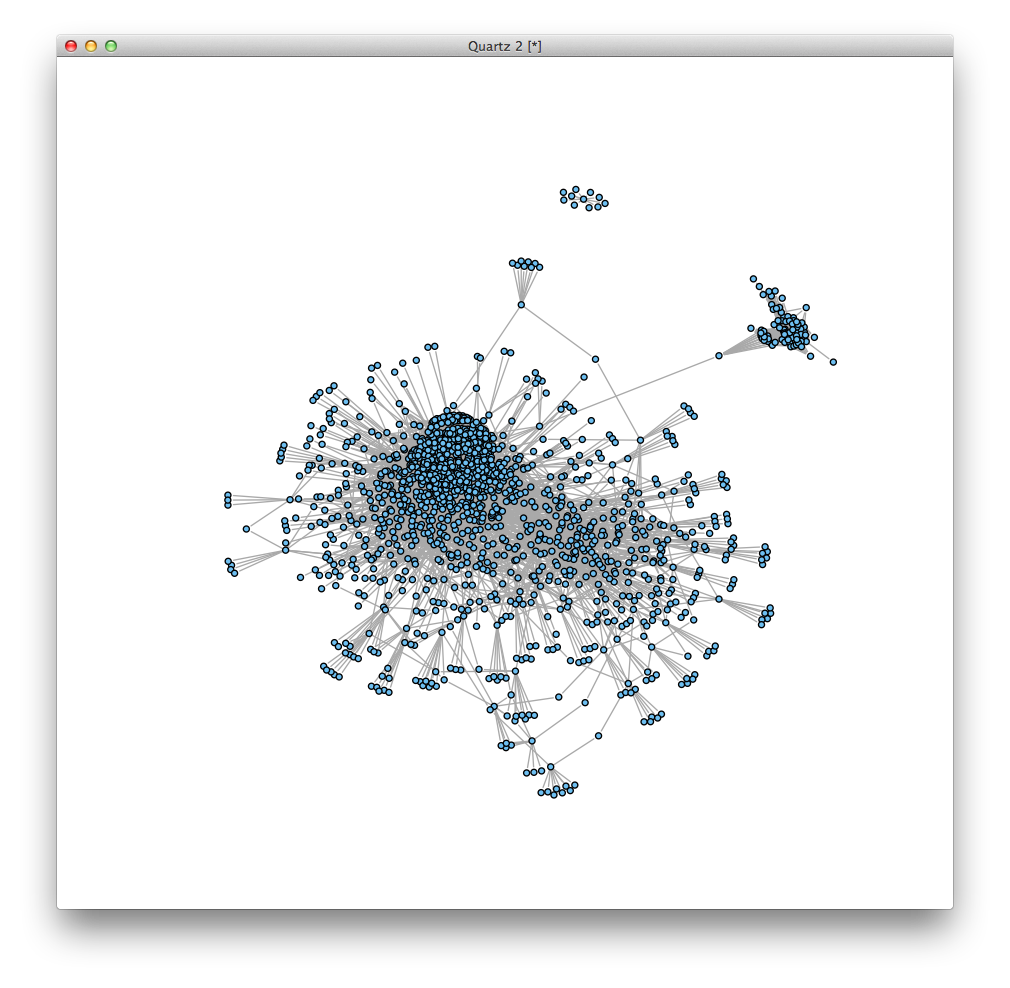
ネットワーク構造の可視化 †
レイアウトをfruchterman.reingoldにして,ネットワークを表示します. ネットワークが大きいので,頂点の大きさ(vertex.size)を2に,頂点のラベル(vertex.label)を非表示(NA)にします.
plot(g,layout=layout.fruchterman.reingold,vertex.size=2,vertex.label=NA)
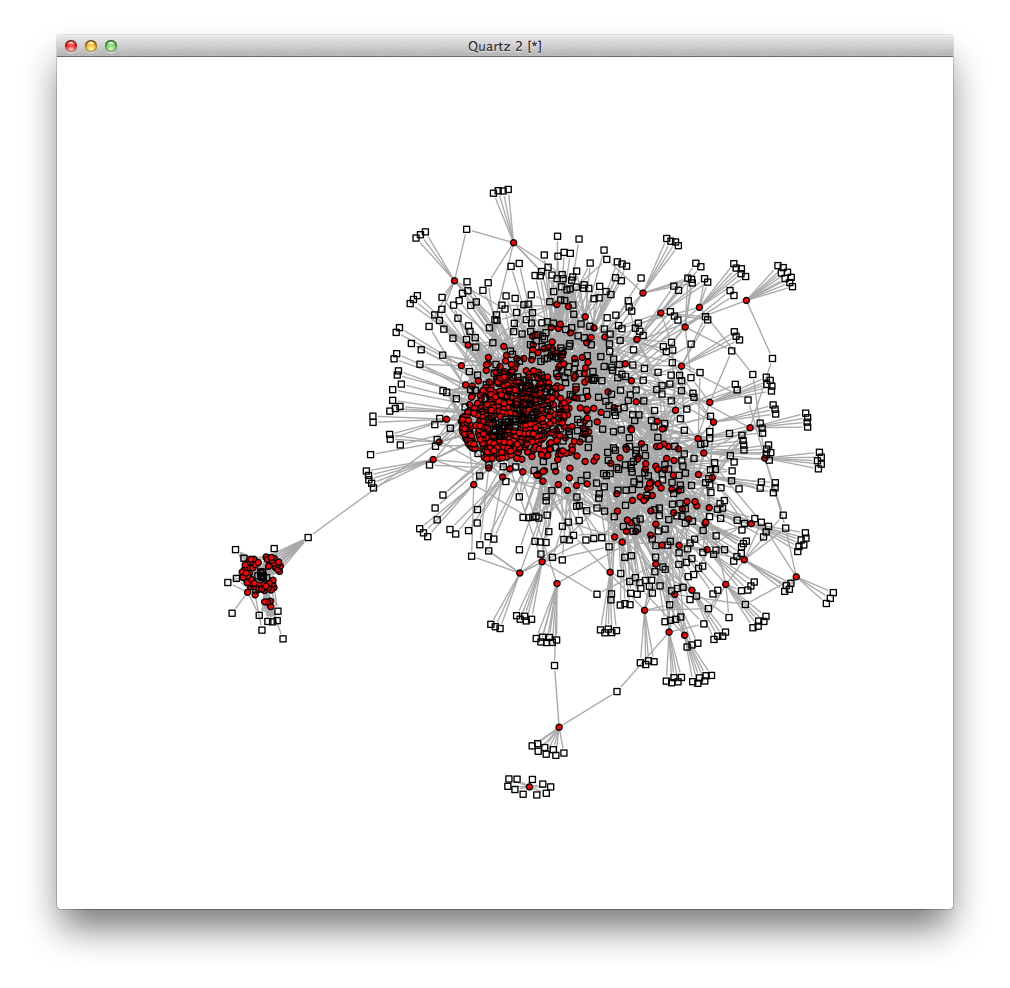
見た目を変える †
このネットワークには1,460個の頂点がありますが,最初の691個と残りの769個は別の種類です. そこで,形と色を変えて表示します.
V(g)$shape="circle" V(g)$color="red" for (i in 692:1460) { V(g)$shape[i]="square" V(g)$color[i]=NA } plot(g,layout=layout.fruchterman.reingold,vertex.size=2,vertex.label=NA)
このfruchterman.reingoldのように,内部で乱数を用いているレイアウトだと表示するたびに形が変わってしまいます. 表示する前に乱数のシードをセットすれば,毎回同じ形で表示されます.
set.seed(0) plot(g,layout=layout.fruchterman.reingold,vertex.size=2,vertex.label=NA)
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)