- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- バイオ・データ・マイニング/Rで回帰分析する へ行く。
- 1 (2011-11-07 (月) 13:30:05)
- 2 (2011-11-07 (月) 16:06:30)
- 3 (2011-11-07 (月) 19:24:10)
- 4 (2011-11-08 (火) 02:31:29)
- 5 (2011-11-08 (火) 10:08:16)
- 6 (2011-11-08 (火) 16:54:20)
- 7 (2011-11-08 (火) 19:54:10)
- 8 (2011-11-10 (木) 15:58:47)
- 9 (2011-11-11 (金) 12:46:43)
- 10 (2011-11-12 (土) 17:00:14)
- 11 (2011-11-15 (火) 08:05:02)
- 12 (2013-11-19 (火) 15:52:23)
- 13 (2013-12-05 (木) 13:03:50)
- 14 (2013-12-05 (木) 13:03:50)
- 15 (2013-12-05 (木) 13:03:50)
- 16 (2016-12-15 (木) 08:40:12)
- 17 (2017-12-14 (木) 20:11:02)
- 18 (2017-12-15 (金) 12:11:37)
- 19 (2017-12-15 (金) 12:11:37)
- 20 (2020-01-07 (火) 19:29:34)
- 21 (2020-01-15 (水) 16:27:42)
はじめに †
『Rによるバイオインフォマティクスデータ解析』の7.17節「LASSO」を参考にして,回帰分析をします.
きちんと理解するために,まずは線形回帰をやります. また,ついでにRidge回帰もやります.
準備 †
まずは,標準で使用できるirisデータを使います.
data(iris)
このデータは,ユリの種類(Species)を花びらの長さ(Sepal.Length),幅(Lepal.Width),がくの長さ(Petal.Length),幅(Petal.Width)によって分類する問題です. 長さと幅は連続値,種類はsetosa, versicolor, virginicaのいずれかをとる離散値です.
150個のデータが含まれています. 最初の10個を表示すると,次のようになります.
iris[sort(sample(1:150,10)),]
ランダムにサンプリングした10個を表示すると,このようになります.
Sepal.Length Sepal.Width Petal.Length Petal.Width Species 4 4.6 3.1 1.5 0.2 setosa 22 5.1 3.7 1.5 0.4 setosa 65 5.6 2.9 3.6 1.3 versicolor 97 5.7 2.9 4.2 1.3 versicolor 100 5.7 2.8 4.1 1.3 versicolor 108 7.3 2.9 6.3 1.8 virginica 116 6.4 3.2 5.3 2.3 virginica 122 5.6 2.8 4.9 2.0 virginica 136 7.7 3.0 6.1 2.3 virginica 146 6.7 3.0 5.2 2.3 virginica
線形回帰(単回帰) †
まず,説明変数が1つの線形回帰(単回帰)を行います. つまり,モデルが次のような式で表される直線をデータから求めます. \[y = \beta_0 + \beta_1 x_1\] ここで,[math]y[/math]が被説明変数(目的変数),[math]x[/math]が説明変数,[math]\beta_0[/math]が定数項,[math]\beta_1[/math]が([math]x[/math]の)係数です.
線形回帰では,訓練データに対する二乗誤差を最小化するような係数および定数項を求めます. つまり,単回帰では,[math]N[/math]個の観測データに対して, \[\sum_{i=1}^N ||y_i - (\beta_0 + \beta_1 x_{i,1})||^2\] を最小化するよう[math]\beta_0[/math]と[math]\beta_1[/math]を求めます. (求め方の説明は省きます.)
ここでは,がくの長さ(Petal.Length)を目的変数,花びらの長さ(Sepal.Length)を説明変数としてモデルを学習しましょう.
この関係をプロットすると次のようになります.
plot(iris$Sepal.Length, iris$Petal.Length)
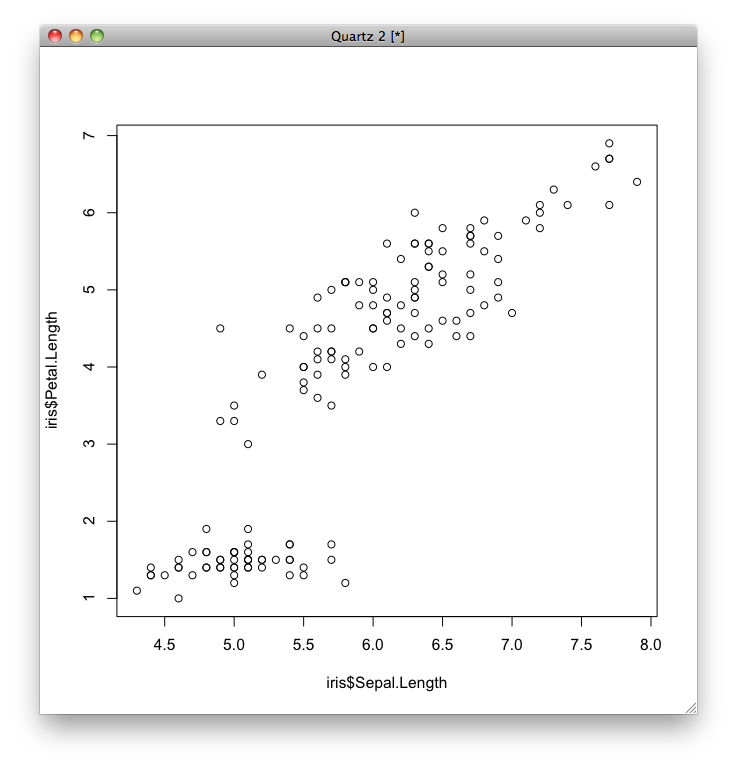
Rでは,モデルの式を,記号~を使って「目的変数 ~ 説明変数」というように表します. したがって,目的変数をがくの長さ(Petal.Length),説明変数を花びらの長さ(Sepal.Length)とするとき,モデルの式は「iris$Petal.Length ~ iris$Sepal.Length」となります.
線形回帰は,標準で用意されているlm関数で行います.
y <- iris$Petal.Length x1 <- iris$Sepal.Length
学習された結果を表示してみると,次のようになります.
iris.sgl <- lm(y ~ x1)
Coefficientsは係数を表し,Interceptは定数項([math]y[/math]切片)の値を表します. つまり,花びらの長さを[math]x[/math],がくの長さを[math]y[/math]とすると,学習された式は次のように書けます. \[y = -7.101443 + 1.858433 x_1\]
学習された線形回帰モデルを図示します.
iris.sgl
すると,次のようなグラフがプロットされます.
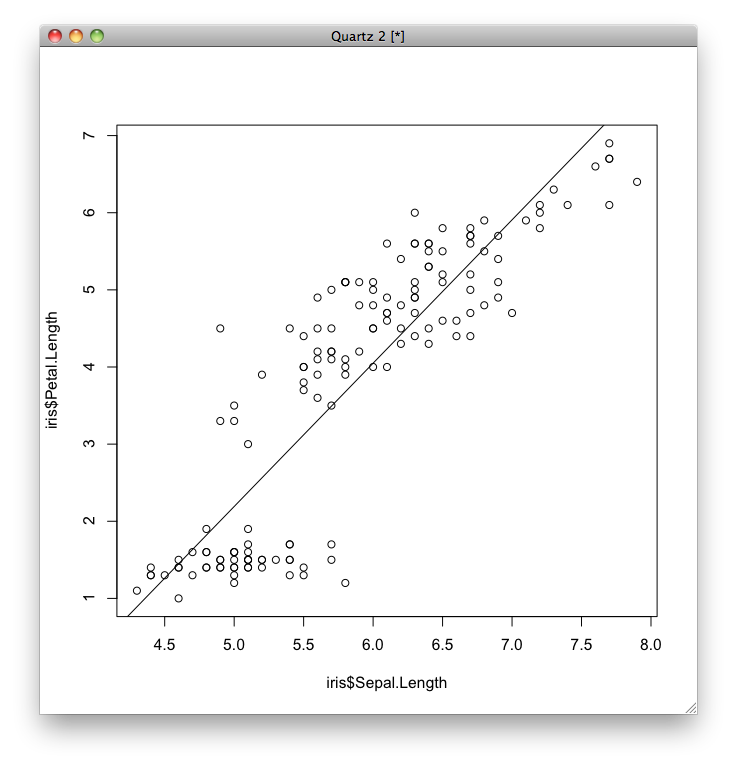
単回帰だとモデルが視覚化しやすいです.
次に,予測誤差を視覚化するため,それぞれのデータについて,横軸に実際のがくの長さ(Petal.Length),縦軸にその予測値をとってグラフを表示します. 予測値はpredict関数で求めることができます.
Call: lm(formula = y ~ x1) Coefficients: (Intercept) x1 -7.101 1.858
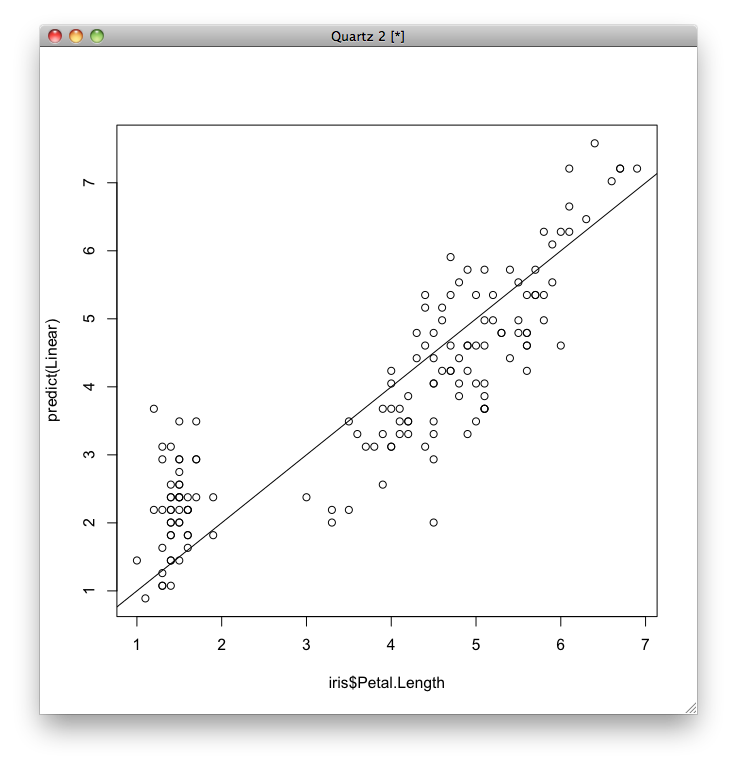
この予測がどのくらい良いのかを表すために,平均二乗誤差(実際の値と予測値の差の二乗の平均)を求めます.
plot(x1, y) abline(iris.sgl)
Meanのところが平均二乗誤差です.
線形回帰(重回帰) †
説明変数が複数ある回帰を重回帰といいます.
重回帰のモデルは次のような式で表されます. \[y = \beta_0 + \beta_1 x_1 + \beta_2 x_2 + \dots + \beta_p x_p\ = \beta_0 + \beta^\mathrm{T}\bf{x}\] ここで,[math]\beta[/math]と[math]\bf{x}[/math]は,それぞれ[math]\beta_1, \dots, \beta_p[/math]と[math]x_1, \dots, x_p[/math]を要素とするベクトルです.
また,単回帰のときと同様に,二乗誤差 \[\sum_{i=1}^N || y_i - (\beta_0 + \beta^\mathrm{T}\bf{x}_i)||^2\] を最小化するような[math]\beta_0, \dots, \beta_p[/math]を求めます.
ここでは,がくの長さ(Petal.Length)を目的変数,花びらの長さ(Sepal.Length),幅(Sepal.Width),がくの幅(Petal.Width)を説明変数として重回帰を行いましょう.
説明変数が複数あるときは,モデルを表すときにそれぞれの変数を記号+で連結して表します. ここでは,「iris$Petal.Length ~ iris$Sepal.Length+iris$Sepal.Width+iris$Petal.Width」となります.
単回帰と同じように,lm関数を用いて線形回帰を行います.
summary(iris.sgl)
これも,花びらの長さ(Sepal.Length)を[math]x_1[/math],幅(Sepal.Width)を[math]x_2[/math],がくの幅(Petal.Width)を[math]x_3[/math],がくの長さ(Petal.Length)を[math]y[/math]とすると,学習された式は次のように書けます. \[y = -0.2627112 + 0.7291384 x_1 - 0.6460124 x_2 + 1.4467934 x_3\]
重回帰分析の場合はモデルが視覚化しにくいので,予測誤差だけ視覚化します.
Call: lm(formula = y ~ x1) Residuals: Min 1Q Median 3Q Max -2.47747 -0.59072 -0.00668 0.60484 2.49512 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) -7.10144 0.50666 -14.02 <2e-16 *** x1 1.85843 0.08586 21.65 <2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Residual standard error: 0.8678 on 148 degrees of freedom Multiple R-squared: 0.76, Adjusted R-squared: 0.7583 F-statistic: 468.6 on 1 and 148 DF, p-value: < 2.2e-16
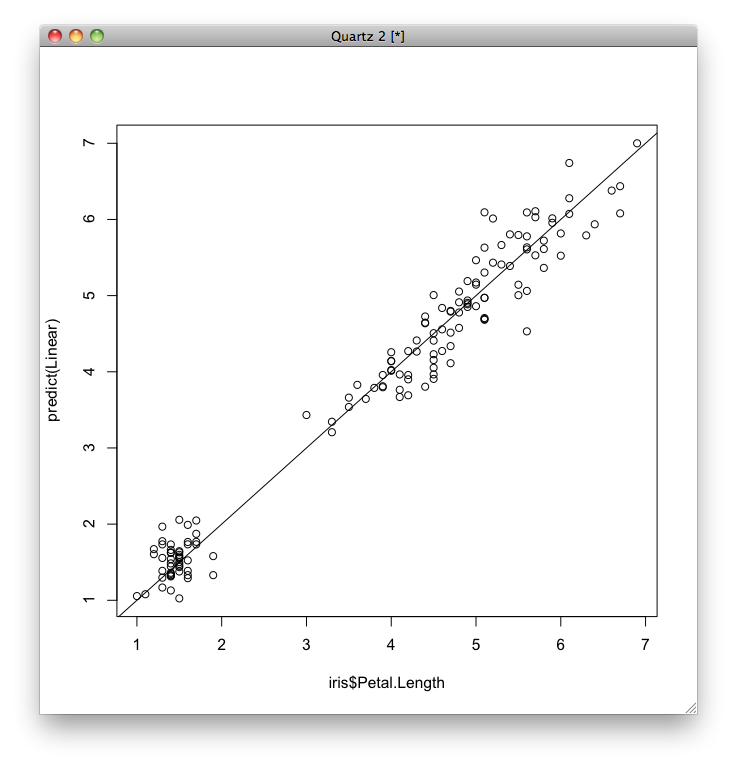
単回帰と同様に,平均二乗誤差を求めます.
plot(y, predict(iris.sgl)) abline(a=0,b=1)
単回帰の場合よりも誤差がかなり小さくなっていることがわかります.
LASSO回帰 †
LASSO回帰は,モデル式は重回帰と同じですが,最小化する目的関数が次のような形をしています. \[\sum_{i=1}^N || y_i - (\beta_0 + \beta^\mathrm{T}\bf{x}_i)||^2 + \lambda |\beta|\] ここで,[math]\lambda|\beta|[/math]を正則化項といい,[math]\lambda[/math]を正則化パラメーターといいます.
LASSO回帰の正則化をL1ノルムといい,次のように計算します. \[|\beta| = \sum_{i=1}^p|\beta_i|\] つまり,L1ノルムは係数の絶対値の和を表します. 正則化パラメーター[math]\lambda[/math]の値が大きいほど0に近い係数を好むようになります.
LASSO回帰はglmnetパッケージに含まれています. glmnetパッケージは そこで,glmnetパッケージをインストールして,使用します.
mean((y - predict(iris.sgl))**2)
私の環境ではこのような警告が出ますが,これは「utilsパッケージのhead関数とtail関数をglmnetパッケージのhead関数とtail関数で置き換えましたよ」という意味なので,気にしなくていいです.
LASSO回帰は,glmnetパッケージのglmnet関数を用いて行います. 引数には,説明変数行列と目的変数(そしてオプション)を与えます.
[1] 0.743061
iris[,-5][,-3]は,irisデータの5列目と3列目を除いたもので,これをas.matrix関数を用いて行列として与えています.
plot関数を使うと,正則化項の大きさとそれぞれの係数の関係をプロットすることができます.
y <- iris$Petal.Length x1 <- iris$Sepal.Length x2 <- iris$Sepal.Width x3 <- iris$Petal.Width
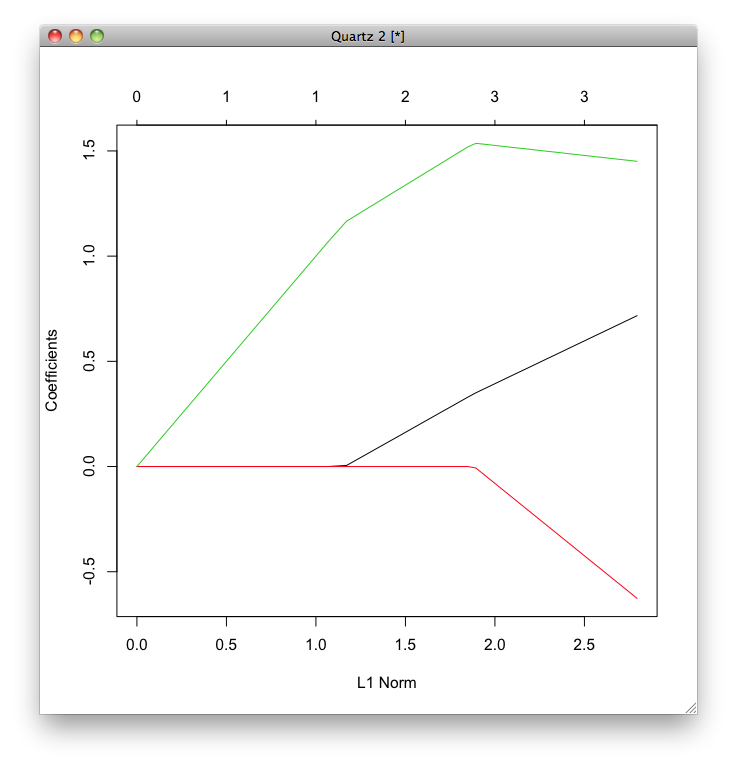
黒色の折れ線が花びらの長さ(Sepal.Length),赤色の折れ線が花びらの幅(Sepal.Width),緑色の折れ線ががくの幅(Petal.Width)を表しています.
縦軸はそれぞれの係数を表しています. 横軸の左端が最も正則化が強いとき,つまり,係数が大きいときのペナルティを最も大きくしたときであり,右端が最も正則化が弱いとき,つまり,係数の大きさに対するペナルティなしのときです.
最も正則化が強いときは,すべての係数が0になっています. また,ペナルティなしのときの係数は,重回帰のときと同じになっています.
次に,最適な正則化パラメーター([math]\lambda[/math])の値を交差検定(クロス・バリデーション)を用いて求めます.
iris.mult <- lm(y ~ x1+x2+x3) iris.mult$coefficients
plotを用いて,[math]\lambda[/math]と平均二乗誤差の関係を表示すると次のようになります.
(Intercept) x1 x2 x3 -0.2627112 0.7291384 -0.6460124 1.4467934
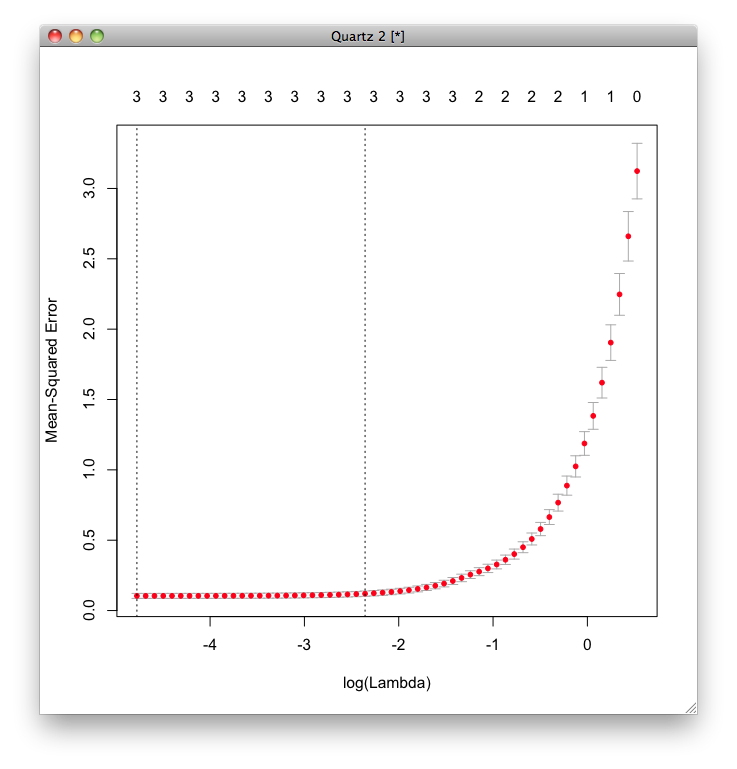
今回の例では正則化パラメーターの値が小さいほうが平均二乗誤差が小さくなりましたが,いつもこうなるわけではありません.
最も予測誤差が小さかったときの[math]\lambda[/math]の値は,クロス・バリデーションによって得られたオブジェクトの変数lambda.minに格納されています.
summary(iris.mult)
係数は変数betaに,定数項の値は変数a0に格納されています. しかし,各[math]\lambda[/math]ごとの係数が格納された行列やベクトルになっていますので,which関数を用いて交差検定の予測誤差が最も小さかったときの[math]\lambda[/math]のインデックスを調べ,そのときの係数と定数項の値だけを表示します.
Call: lm(formula = y ~ x1 + x2 + x3) Residuals: Min 1Q Median 3Q Max -0.99333 -0.17656 -0.01004 0.18558 1.06909 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) -0.26271 0.29741 -0.883 0.379 x1 0.72914 0.05832 12.502 <2e-16 *** x2 -0.64601 0.06850 -9.431 <2e-16 *** x3 1.44679 0.06761 21.399 <2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Residual standard error: 0.319 on 146 degrees of freedom Multiple R-squared: 0.968, Adjusted R-squared: 0.9674 F-statistic: 1473 on 3 and 146 DF, p-value: < 2.2e-16
重回帰と同様に表すと,学習された式を次のように書くことができます. \[y = -0.2557475 + 0.7171234 x_1 - 0.6267432 x_ 2 + 1.4504055 x_3\]
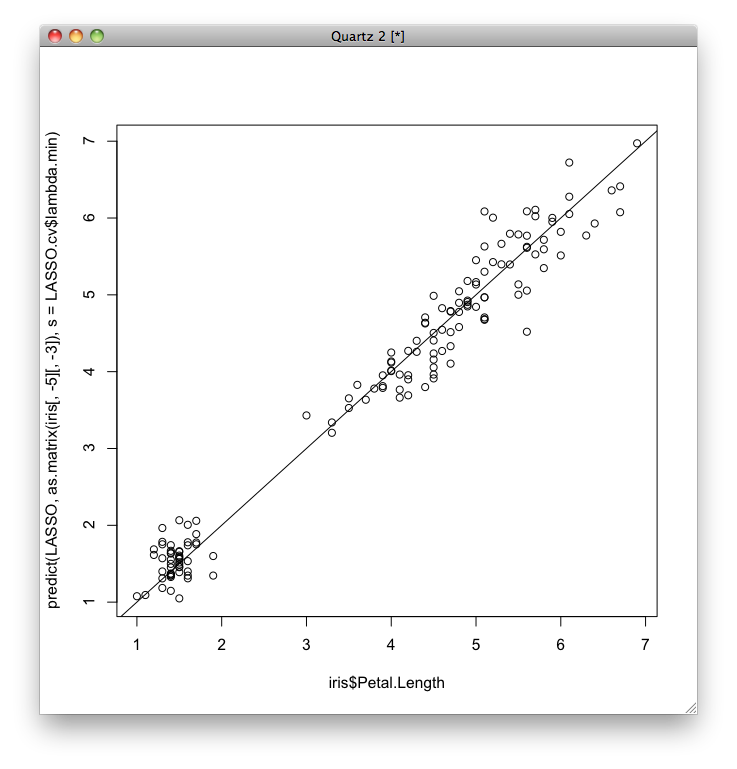
単回帰,重回帰のときと同じように,平均二乗誤差を求め,予測誤差を視覚化します.
plot(y, predict(iris.mult)) abline(a=0,b=1)
Ridge回帰 †
Ridge回帰は,目的関数の正則化項が[math]\lambda||\beta||_2^2[/math]となっている回帰です. つまり,Ridge回帰の目的関数は次のような形をしています. \[\sum_{i=1}^N || y_i - (\beta_0 + \beta^\mathrm{T}\bf{x}_i)||^2 + \lambda ||\beta||_2^2\]
Ridge回帰の正則化をL2ノルムといい,次のように計算します. \[||\beta||_2^2 = \sum_{i=1}^p \beta_i^2\]
LASSO回帰では正則化項のペナルティが係数の絶対値に比例して増加しましたが,Ridge回帰では係数の2乗に比例して増加します.
Ridge回帰を行うには,glmnet関数で学習する際にオプションでalpha=0を指定します.
mean((y - predict(iris.mult))**2)
後はLASSO回帰のときと同じです.
まず,正則化項の大きさと係数の関係を表示します.
[1] 0.09901965
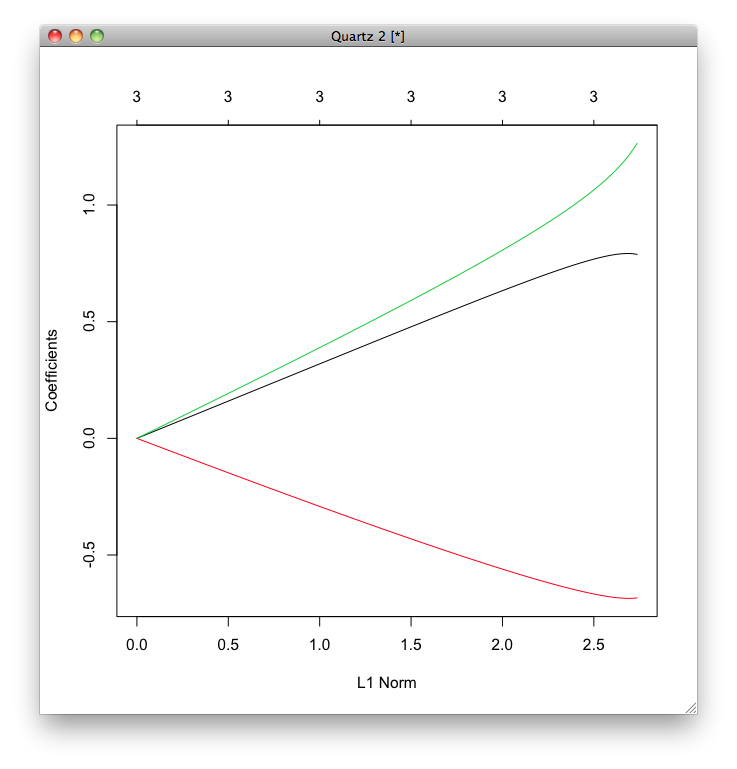
LASSO回帰のグラフと比べると,LASSO回帰の方が係数を0にする傾向が強いことがわかります.
次に,クロス・バリデーションによって最適な[math]\lambda[/math]を求め,そのときの係数と定数項を表示します.
install.packages("glmnet") library(glmnet)
これまでと同様に表すと,学習された式は次のように書けます. \[y = -0.2742464 + 0.7882905 x_1 - 0.6838069 x_2 + 1.2645594 x_3\]
最後に,平均二乗誤差を求め,予測誤差をプロットします.
x <- cbind(x1, x2, x3) iris.lasso <- glmnet(x, y)
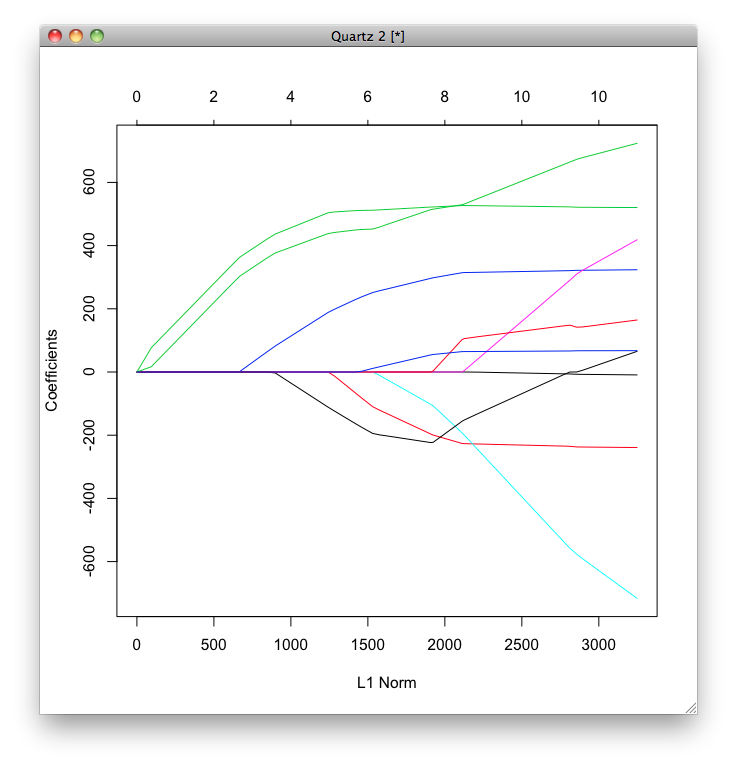
ちなみに,LASSO回帰のときと同じように[math]\lambda[/math]と平均二乗誤差の関係をプロットしようとしましたが,できませんでした.
plot(iris.lasso)
応用 †
diabetes(糖尿病)データに応用してみましょう.
このデータはlarsパッケージ*1に含まれていますので,まずはlarsパッケージをインストールします.
iris.lasso.cv = cv.glmnet(x, y)
diabetesデータには,442個の訓練データが含まれており,10次元の説明変数を表す行列xと目的変数を表すベクトルyが格納されています.
まず,重回帰を行い,平均二乗誤差を求めます.
plot(iris.lasso.cv)
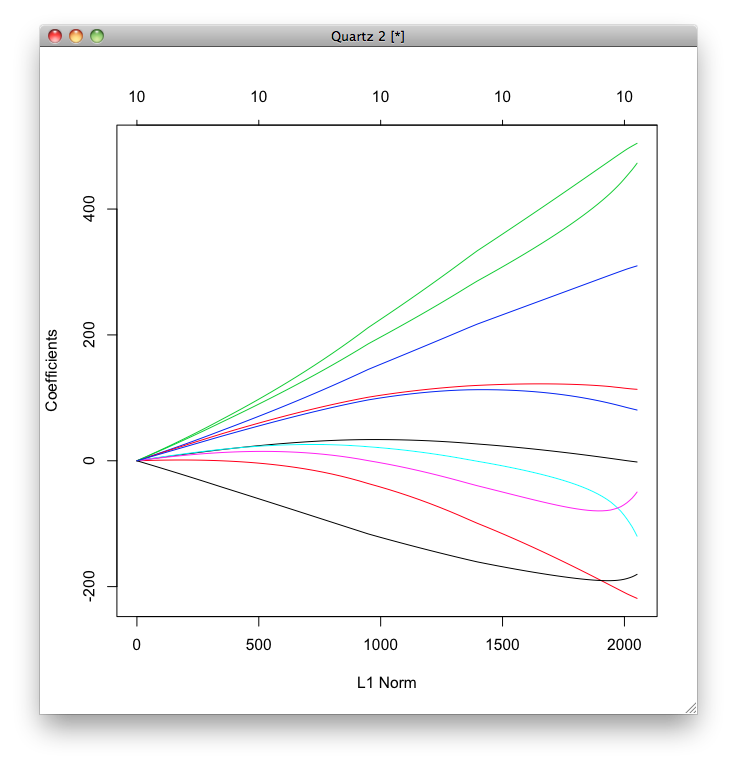
次に,LASSO回帰を行い,正則化項と係数の関係をプロットします.
iris.lasso.cv$lambda.min
正則化が弱いときは(つまり,グラフの右の方では),irisに比べて係数がかなり大きいことがわかります.
クロス・バリデーションでLASSO回帰の最適な[math]\lambda[/math]を求めます.
[1] 0.008431423
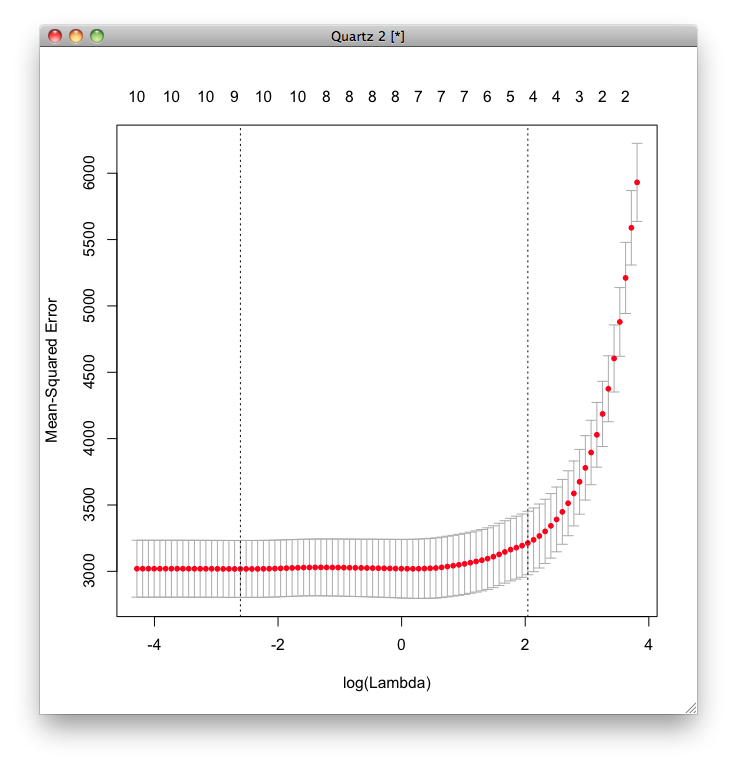
LASSO回帰の平均二乗誤差を求めます.
iris.lasso$beta[,which(iris.lasso$lambda==iris.lasso.cv$lambda.min)]
最後に,Ridge回帰を行います.
Sepal.Length Sepal.Width Petal.Width 0.7171234 -0.6267432 1.4504055
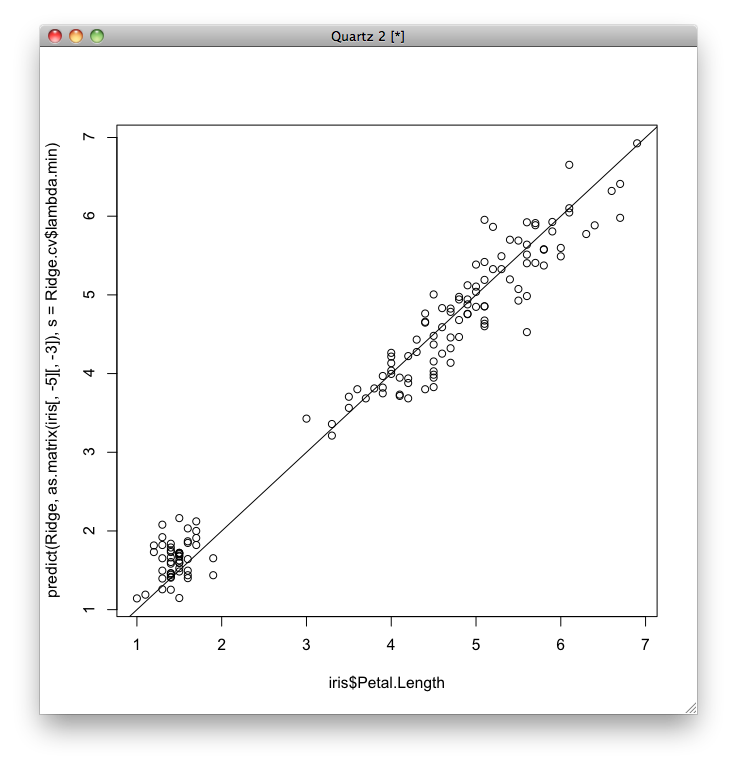
クロス・バリデーションによってRidge回帰の最適な[math]\lambda[/math]を求めます.
iris.lasso$a0[which(iris.lasso$lambda==iris.lasso.cv$lambda.min)]
Ridge回帰の平均二乗誤差を求めます.
s57 -0.2557475
まとめ †
LASSO回帰やRidge回帰は,誤差関数に正則化項を加えることによってモデルに制約を加えるものです.
参考文献 †
7.17節「LASSO」を理解するためにこのページを作成しました.
LASSO回帰とRidge回帰については,この本に詳しく載っています.
注 †
この記事はまだ書きかけです.
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)