- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- バイオ・データ・マイニング/HMMERで相同性検索を行う へ行く。
はじめに †
ここでは,HMMERを使って相同性検索を行います.
相同性検索 †
相同性検索については次のページで説明しています.
- BLASTで相同性検索を行う - とうごろうぃき
HMMERによる相同性検索を行う前に,BLASTによる相同性検索を先に理解しておくことをおすすめします.
HMMER †
HMMERはモチーフをクエリーとして相同性検索を行うツールです. (反対に配列をクエリーとしてモチーフを検索することもできますが,ここでは説明しません.)
モチーフとは,タンパク質のドメインを構造化したものです.
タンパク質のドメインは,タンパク質のアミノ酸配列の部分配列のうち,タンパ質の機能を発現するのに必要な部分です. 同じ機能をもつ(異なる種の)タンパク質には,同じ(あるいは,とても良く似た)ドメインが含まれています. ドメイン以外の部分は大きく異なっていても,ドメインが同じであれば(あるいは,とても良く似ていれば )同じ機能を発現します.
HMMERでは,モチーフはプロファイルHMMで表現されます.
プロファイルHMM †
プロファイルHMMは,多樹配列アラインメントの結果を一つにまとめたものであるプロファイルを隠れマルコフモデル (HMM, hidden Markov model) で表したものです.
状態遷移確率が直前の状態のみに依存するというマルコフ性を仮定して状態遷移確率をモデル化したものがマルコフ・モデルであり,マルコフ・モデルにおいて状態が観測できず(隠れていて)出力だけが観測できるものがHMMです.
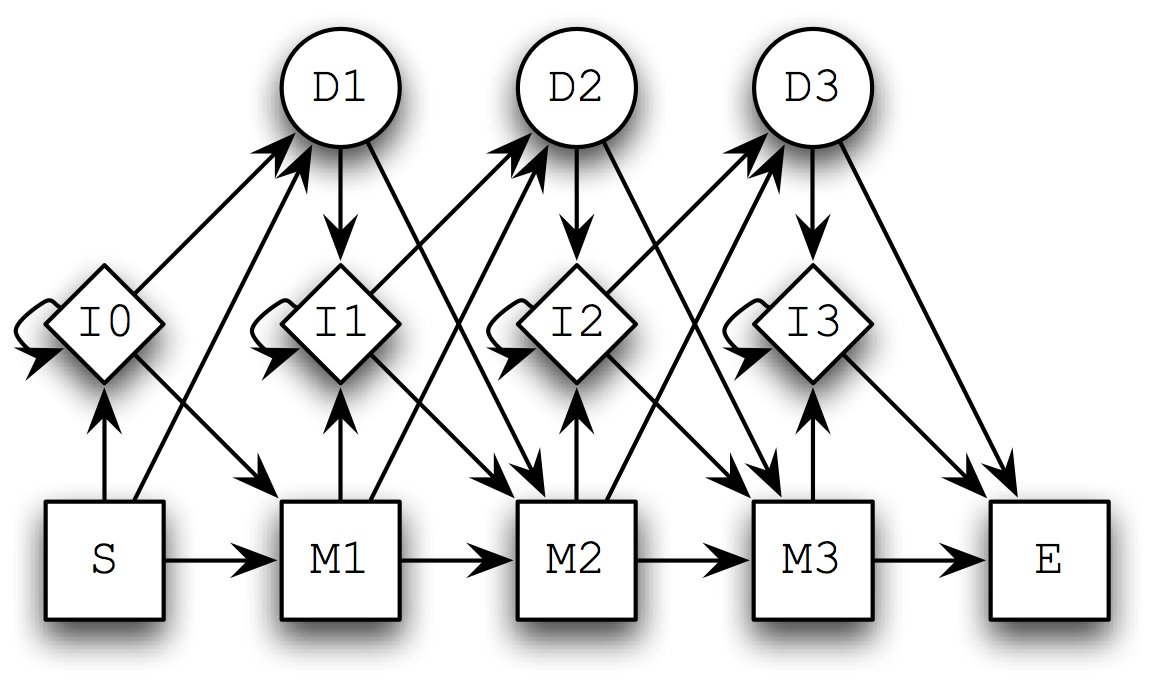
たとえば, 長さ3のドメインのHMMは次のようなものになります.
Sが開始状態,Eが終端状態,Mが一致状態,Iが挿入状態,Dが削除状態を表します. 出力は,一致状態と挿入状態ではアミノ酸のいずれか,削除状態では出力なし(ギャップだけど明示的には観測できない)となります.
以下のような多重アラインメントで表されるモチーフのプロファイルHMMを考えてみましょう.
foo CD-F
bar CDEF
baz C--Y
* :
最初のアミノ酸は C ですべて一致していますので,[math]\Pr(M_1|S) = 1[/math] となります.(以下,明示的に書かないところは確率ゼロです.) また,[math]M_1[/math] での出力確率は [math]\Pr(C|M_1) = 1[/math] となります.
次のアミノ酸は3本中2本が D なので,[math]\Pr(M_2|M_1) = 2/3[/math], [math]\Pr(D_2|M_1) = 1/3[/math] となります. 出力確率は [math]\Pr(D|M_2) = 1[/math] です.
その次のアミノ酸は1本に E が挿入されているので,[math]\Pr(I_2|M_2) = 1/2[/math], [math]\Pr(M_3|M_2) = 1/2[/math], [math]\Pr(M_3|D_2) = 1[/math] となります. [math]I_2[/math] での出力確率は [math]\Pr(E|I_2) = 1[/math] であり,[math]M_3[/math] での出力確率は [math]\Pr(F|M_3) = 2/3[/math], [math]\Pr(Y|M_3) = 1/3[/math] です.
HMMERに含まれるhmmbuildというツールを使って,ClustalWの多重アラインメントの出力からプロファイルHMMを作ることができます.
プロファイルHMMは,EMアルゴリズムの一種であるBaum-Welchアルゴリズムによって作られます.
また,Viterbiアルゴリズムを用いて,プロファイルHMMに最も一致するアミノ酸配列を出力することができます.
タンパク質のファミリー †
タンパク質のファミリーとは,進化的祖先が同じタンパク質,つまり相同体を集めたものです.
進化的祖先が同じファミリーを集めたものをスーパーファミリーといい,あるスーパーファミリーに含まれるファミリーをそのスーパーファミリーのサブファミリーといいます.
たとえば,ヘモグロビン (Hemoglobin) はグロビン (Globin) というファミリーに属しています.
タンパク質が所属しているファミリーは,UniProtのFamily & Domainsで確認できます.
プロファイルHMMデータベース †
タンパク質の機能が確認されているドメインのモチーフをプロファイルHMMにして,データベースにしたものが Pfamです.
Pfamでは,アミノ酸配列をクエリーとして,クエリー配列に一致するモチーフ(プロファイルHMM)を検索することができます.
ここでは,UniProtに登録されているファミリー情報 (Family & Domains) からPfamへのリンクをたどることで,プロファイルHMMを入手します.
HMMERによるタンパク質の相同性検索 †
PfamからプロファイルHMMを取得して,HMMERで相同性検索を行います.
プロファイルHMMを取得する †
まず,UniProtでタンパク質を検索し,Family & Domainsを表示します.
#ref(): File not found: "uniprot_hba_human.png" at page "バイオ・データ・マイニング/HMMERで相同性検索を行う"
つぎに,Family and domain databasesにあるPfamへのリンクをクリックします.
#ref(): File not found: "uniprot_hba_human_family_and_domains.png" at page "バイオ・データ・マイニング/HMMERで相同性検索を行う"
すると,Pfamのページに移動しますので,ここで,Curation & modelを表示します.
#ref(): File not found: "pfam_globin.png" at page "バイオ・データ・マイニング/HMMERで相同性検索を行う"
一番下にプロファイルHMMについての情報が載っていて,ファイルをダウンロードできます.
#ref(): File not found: "pfam_globin_model.png" at page "バイオ・データ・マイニング/HMMERで相同性検索を行う"
HMMERを実行する †
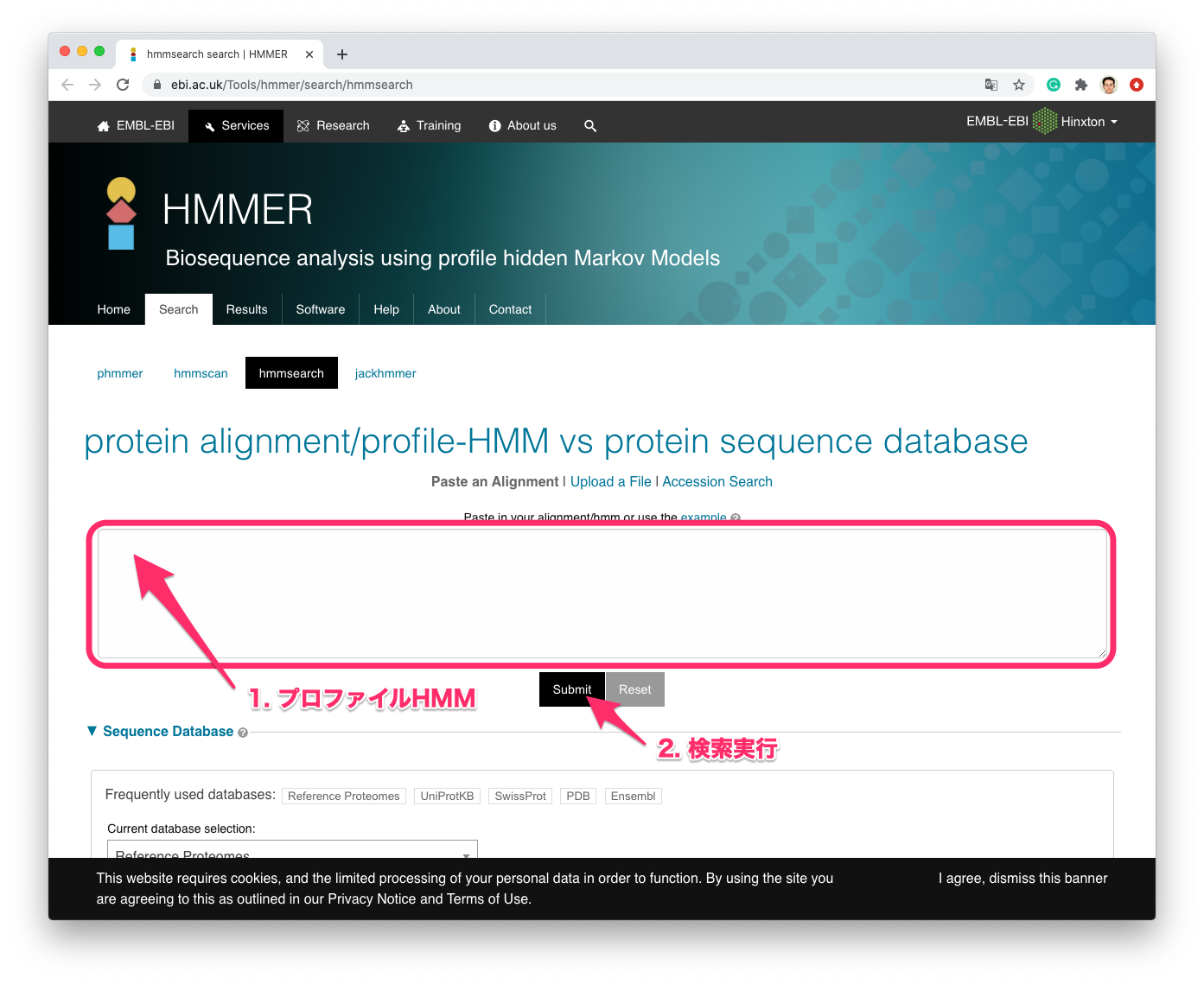
プロファイルHMMをクエリーとしてタンパク質のアミノ酸配列を検索するには,hmmsearchを使います.
HMMERのhmmsearchのテキスト・エリアにダウンロードしたプロファイルHMMのテキストをすべてコピー・アンド・ペーストするか,Upload a Fileでダウンロードしたファイルを指定して,Submitボタンを押します.
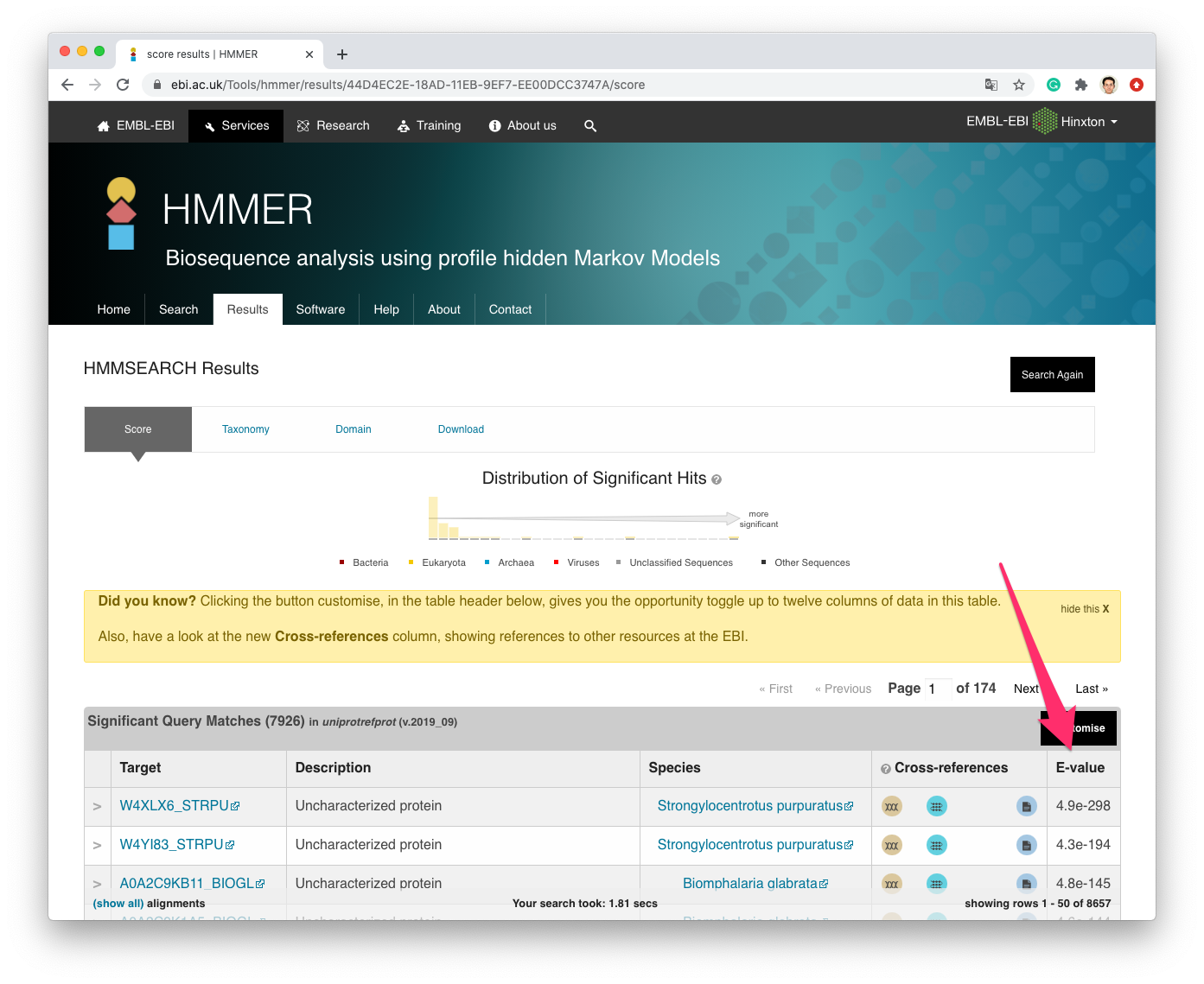
すると,検索結果が表示されます. 最初の画面では,スコア (E-value) が小さい順にタンパク質が並んでいます.
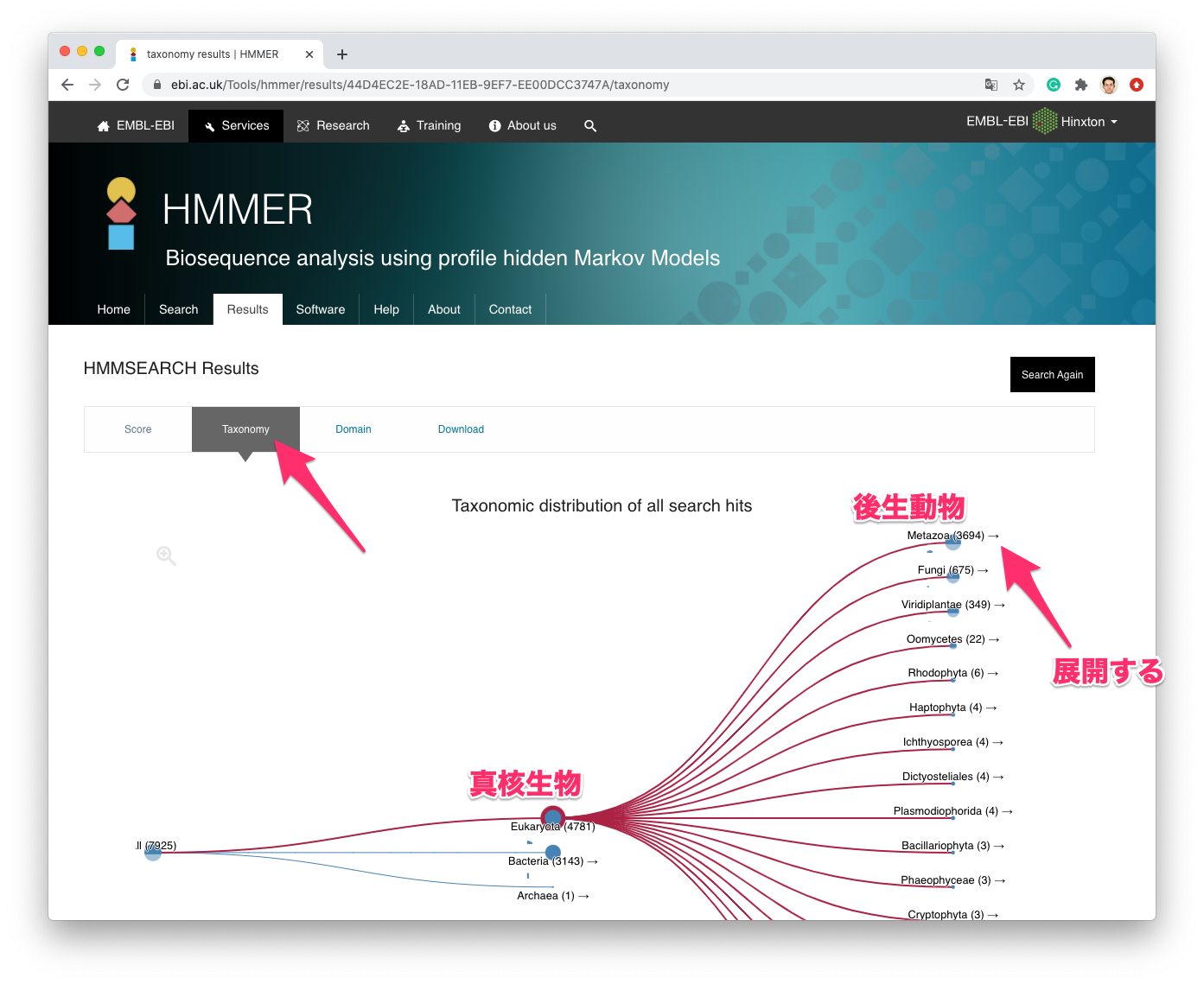
Taxonomyをクリックすると,種ごとに分類されて表示されます.
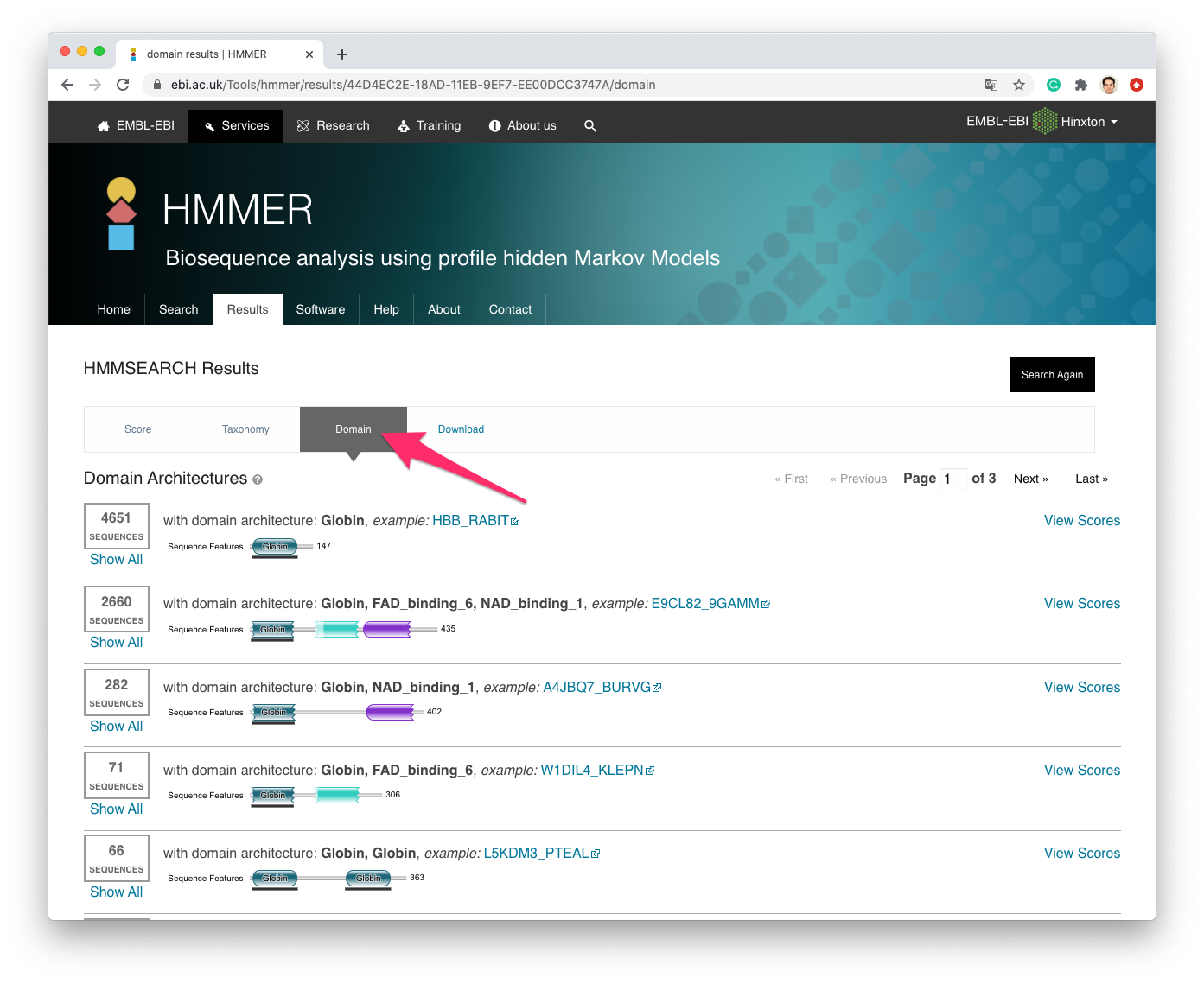
Domainをクリックすると,Pfamのドメインごとに分類されて表示されます.
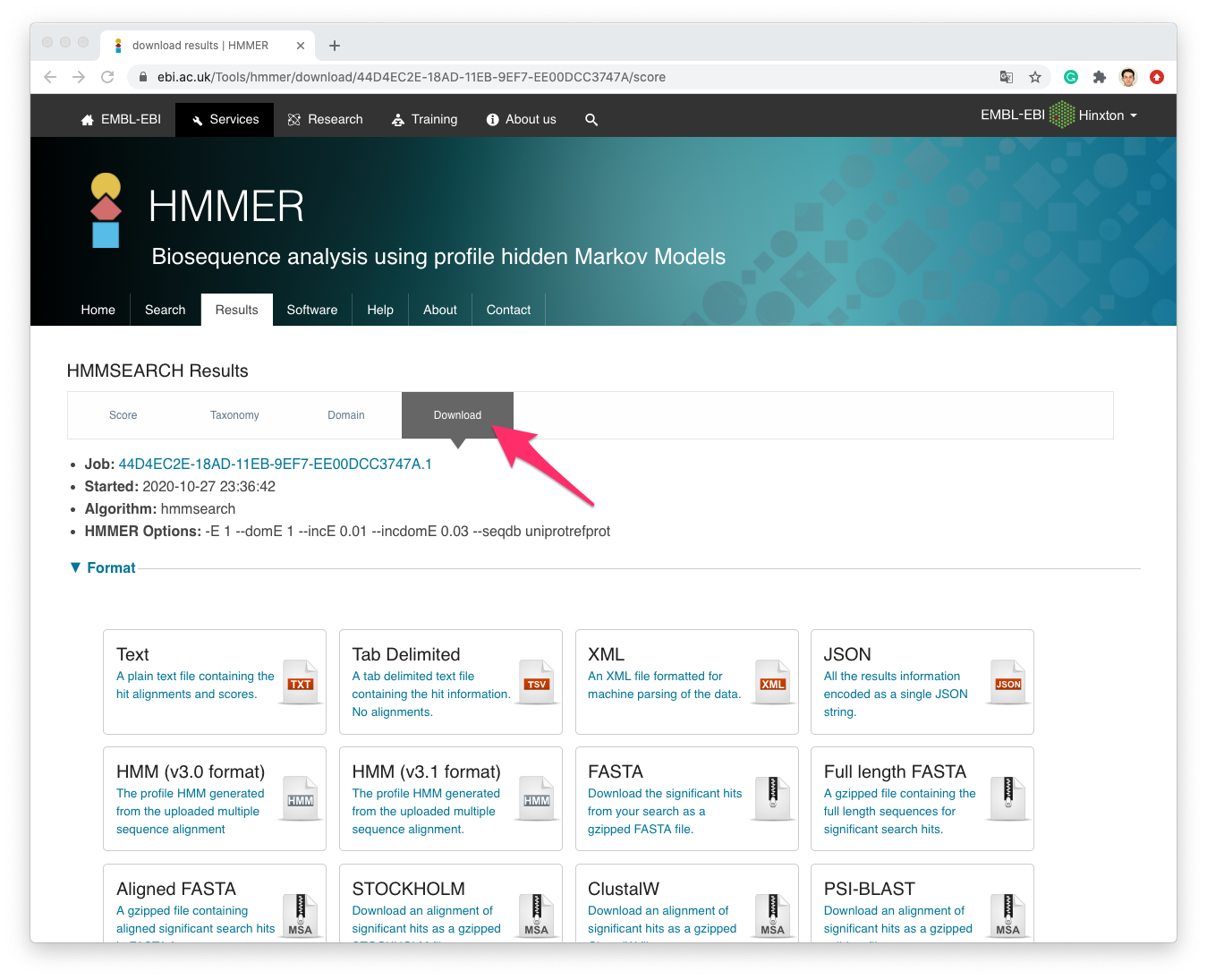
Downloadをクリックすると,検索結果をいろいろなフォーマットでダウンロードできます.
演習 †
UniProtからヒトのアミラーゼ AMY1_Human が所属するファミリーを調べ,PfamからそのファミリーのプロファイルHMMを入手し,HMMERを用いて相同性検索を行ってみよう.
参考文献 †
- 6章 類似配列のデータベース検索
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)