- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- バイオ・データ・マイニング/BLASTで相同性検索を行う へ行く。
はじめに †
ここでは,BLASTを使って相同性検索を行います.
相同性検索 †
DNAやタンパク質が進化的に共通の祖先を持つものを相同体 (homologue) といい,DNAやタンパク質が進化的に共通の祖先を持つことを相同性 (homology) といいます.
DNAデータベースの中からクエリーとして与えられた塩基配列の相同体を見つける,あるいは,タンパク質データベースの中からクエリーとして与えられたアミノ酸配列の相同体を見つけることを相同性検索といいます.
BLAST †
BLASTは配列をクエリーとして相同性検索を行うツールです.
BLASTには,DNAの塩基配列をクエリーとして塩基配列を検索するblastn,タンパク質のアミノ酸配列をクエリーとしてアミノ酸配列を検索するblastp,DNAの塩基配列をクエリーとして(それを翻訳して)アミノ酸配列を検索するblastxなどが含まれています.
ここでは,タンパク質のアミノ酸配列をクエリーとしてアミノ酸配列を検索するblastpを対象にします.
BLASTによる相同性検索の仕組み †
BLASTは,まず,クエリーとして与えられたタンパク質のアミノ酸配列を3文字ずつ(DNAの塩基配列9文字分相当)に分割します. 情報科学の分野では n-gram と呼ばれている技術の n=3 のときと同じです.
たとえば,CDEFGHI というクエリー配列が与えられると,これを CDE, DEF, EFG, FGH, GHI という長さ3の部分文字列に分解します.
次に,アミノ酸置換確率行列に基づいて,生成した部分文字列の類似文字列を作成します.
たとえば,アミノ酸 I はアミノ酸 V と類似している(置換される確率が高い)とき,GHI の I を V に置換したGHV を類似文字列として作成します.
部分文字列または類似文字列に一致する個所を,タンパク質データベースのアミノ酸配列の中から探し,一致した度合いに基づいて評価値 E-value を計算します. 部分文字列が一致するかどうかには有限オートマトンが使われています.
E-value †
検索対象のデータベースにおいて,クエリー配列が偶然に一致するタンパク質の数の期待値を E-value といいます.
まず,検索対称のタンパク質データベースに出現するアミノ酸の数をカウントします. これによって,各アミノ酸が独立に出現すると仮定したときの出現確率が求まります. ここから,各アミノ酸が独立に出現すると仮定したときにあるアミノ酸配列が偶然に生起する確率が求まります.
各アミノ酸が独立に出現するときにランダムに生成したアミノ酸配列のアラインメントのスコアの分布は,Gumbel分布(極値分布のひとつ)になることが分かっています. そこで,Gumbel分布の上でスコアが [math]S[/math] 以上になる確率を求め,それにクエリー配列の長さ [math]m[/math] とデータベースのサイズ [math]n[/math] を乗じることによって,ランダムに生成されたスコア [math]S[/math] 以上のアミノ酸配列の数の期待値を求めます(実際には手順が違います).
これが E-value です.
したがって,同じタンパク質データベースの中で比較すると, E-valueの値は小さい方が良い(偶然に一致するタンパク質の数少ない)です.
タンパク質データベース †
検索対象となるタンパク質データベースは,いくつかの種類があります.
nr は,Non-redundant を表し,色々なデータベースを統合し,重複を取り除いたものです.
refseq は,Reference sequenceを表し,NCBI(アメリカの国立バイオテクノロジー情報センター)がチェックしてから登録しているリファレンスとなる代表的なアミノ酸配列のデータベースです.
SwissProt や PDB や Patent は,それぞれ独自に構築されているタンパク質データベースです. SwissProtは幅広い知識を注釈としてつけたもの,PDBはタンパク質の立体構造で分類したもの,Patentは特許出願に含まれているタンパク質を集めたものです.
BLASTによるタンパク質の相同性検索 †
タンパク質データベースの一つであるUniProtからタンパク質のアミノ酸配列を取得して,BLASTで相同性検索を行います.
UniProtからアミノ酸配列を取得する方法については,以下のページを参照してください.
ここでは,ヒトのヘモグロビン HBA_HUMAN のアミノ酸配列をクエリーとして,NCBIのBLASTで,refseq を対象にして検索してみます.
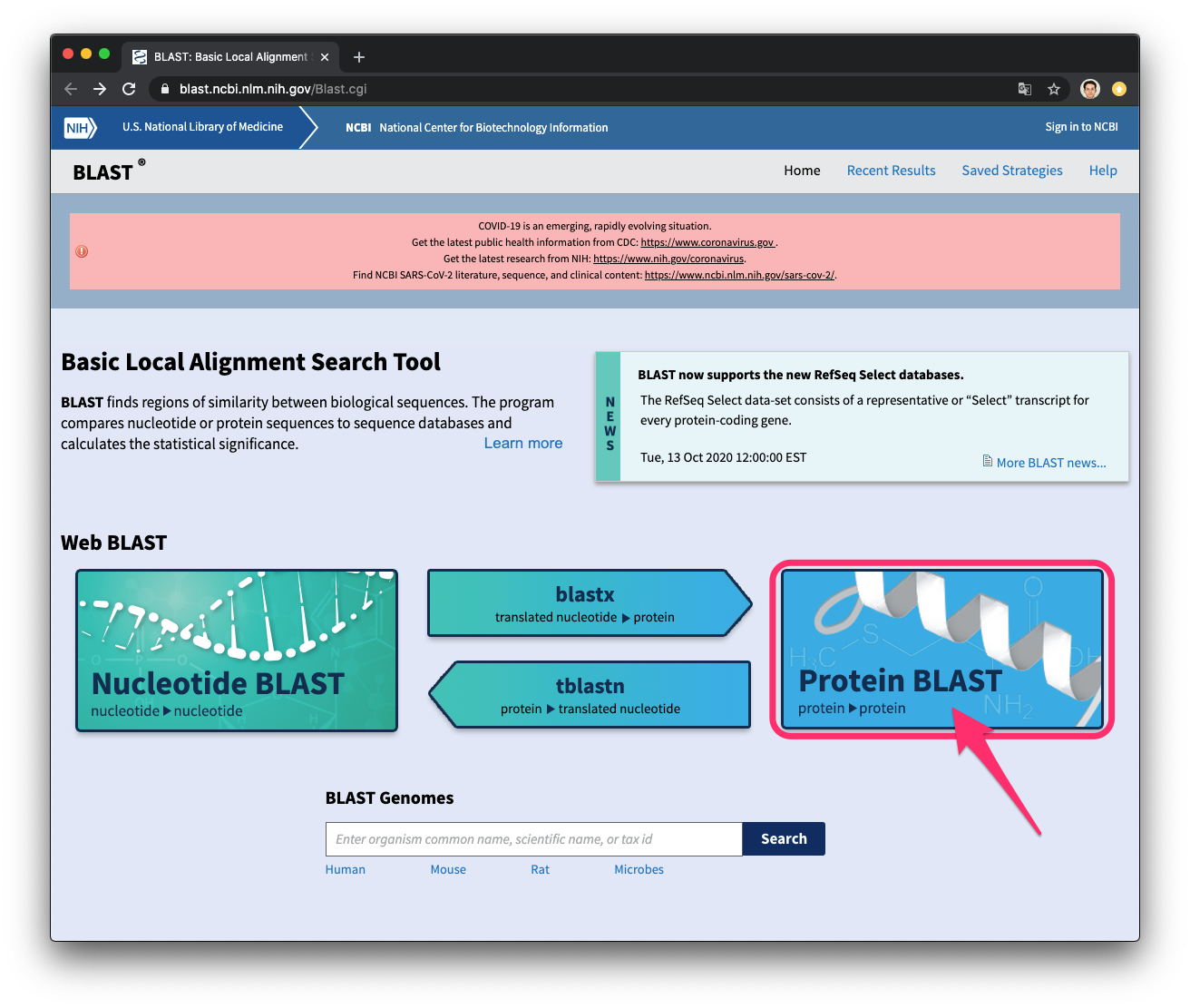
まず,protein blastを選択します.
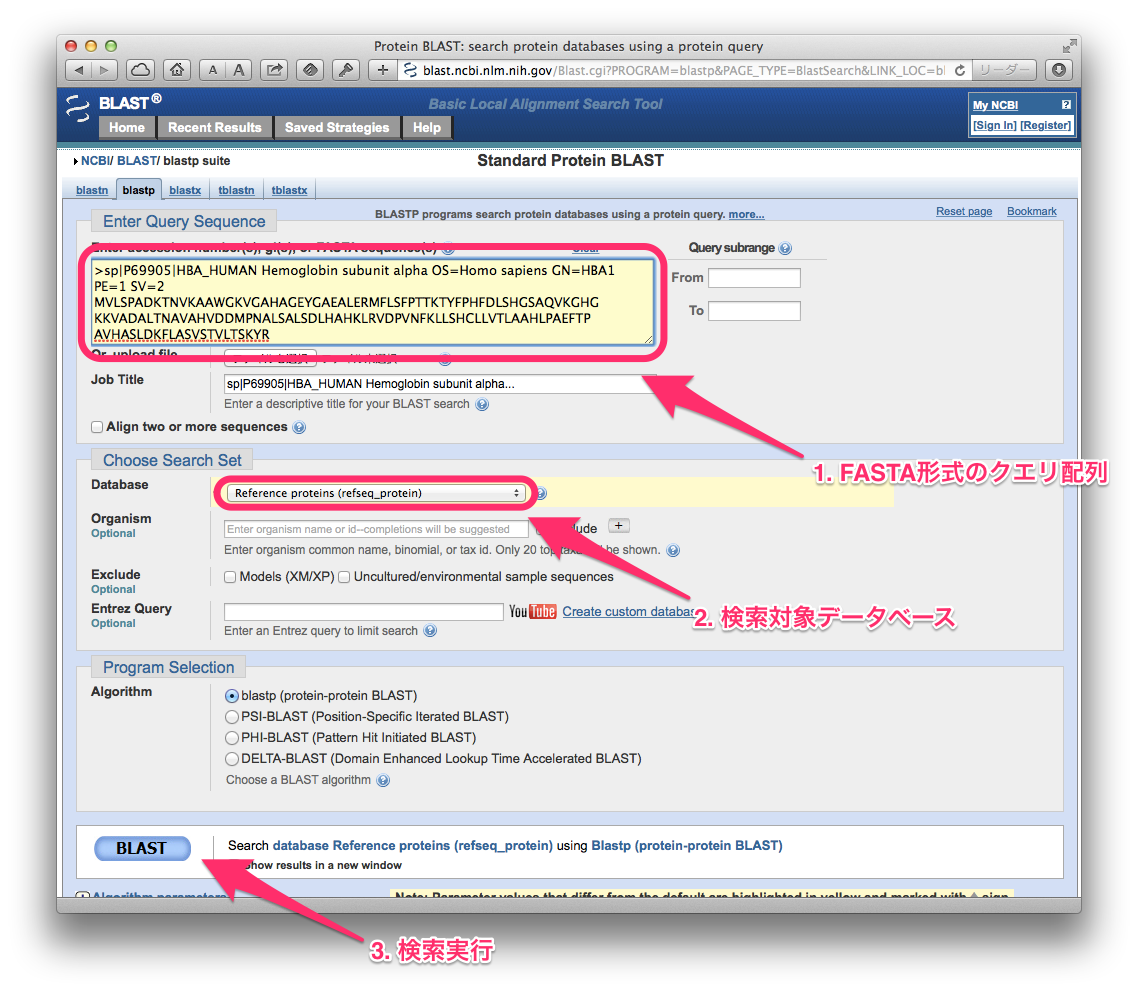
次に,クエリーのところにUniProtから入手したFASTA形式の配列をコピー・アンド・ペーストし,検索対象データベースを選択し,検索実行ボタンを押します.
すると,検索が実行されますが,時間がかかるので,次のような画面が表示され,2秒ごとに更新されます.
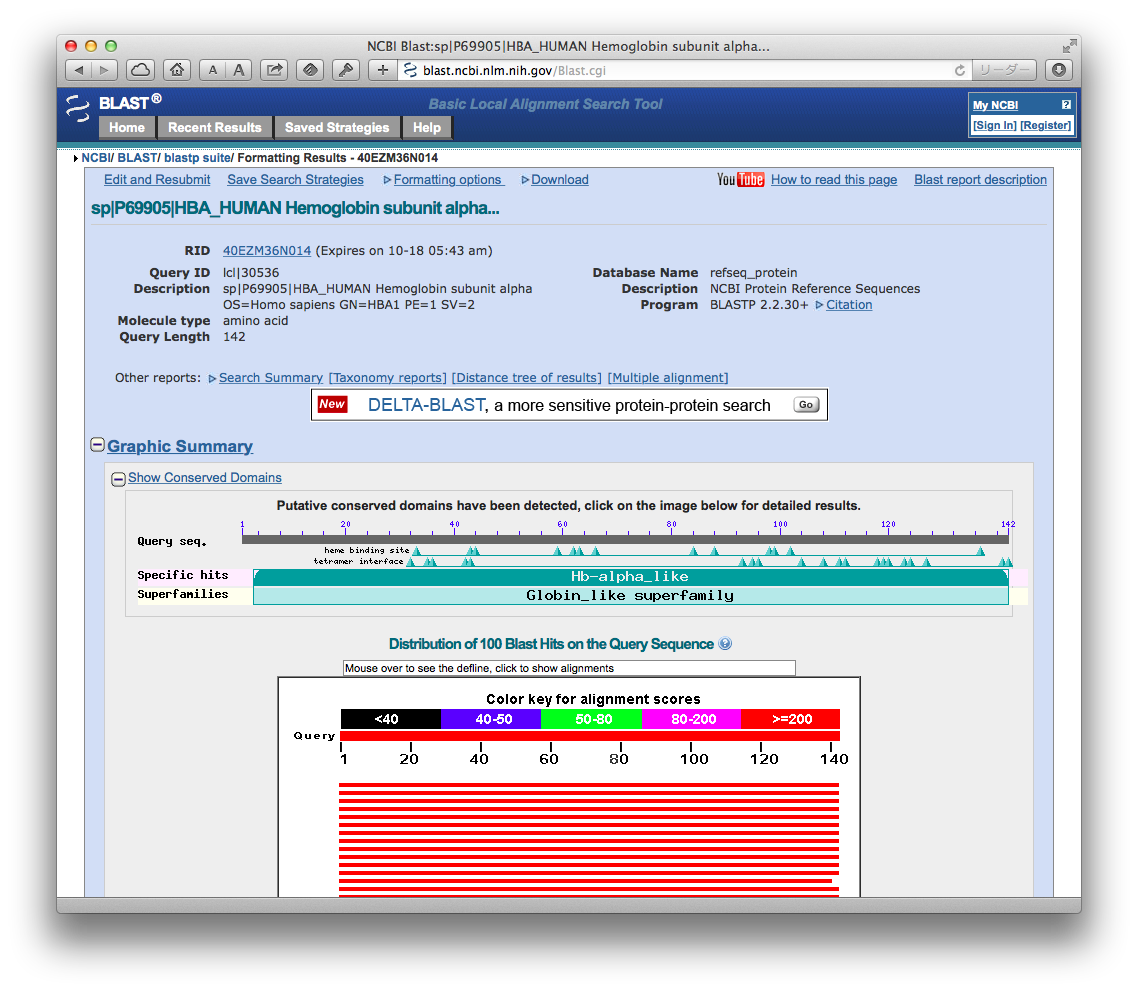
検索が終わると,結果が表示されます.
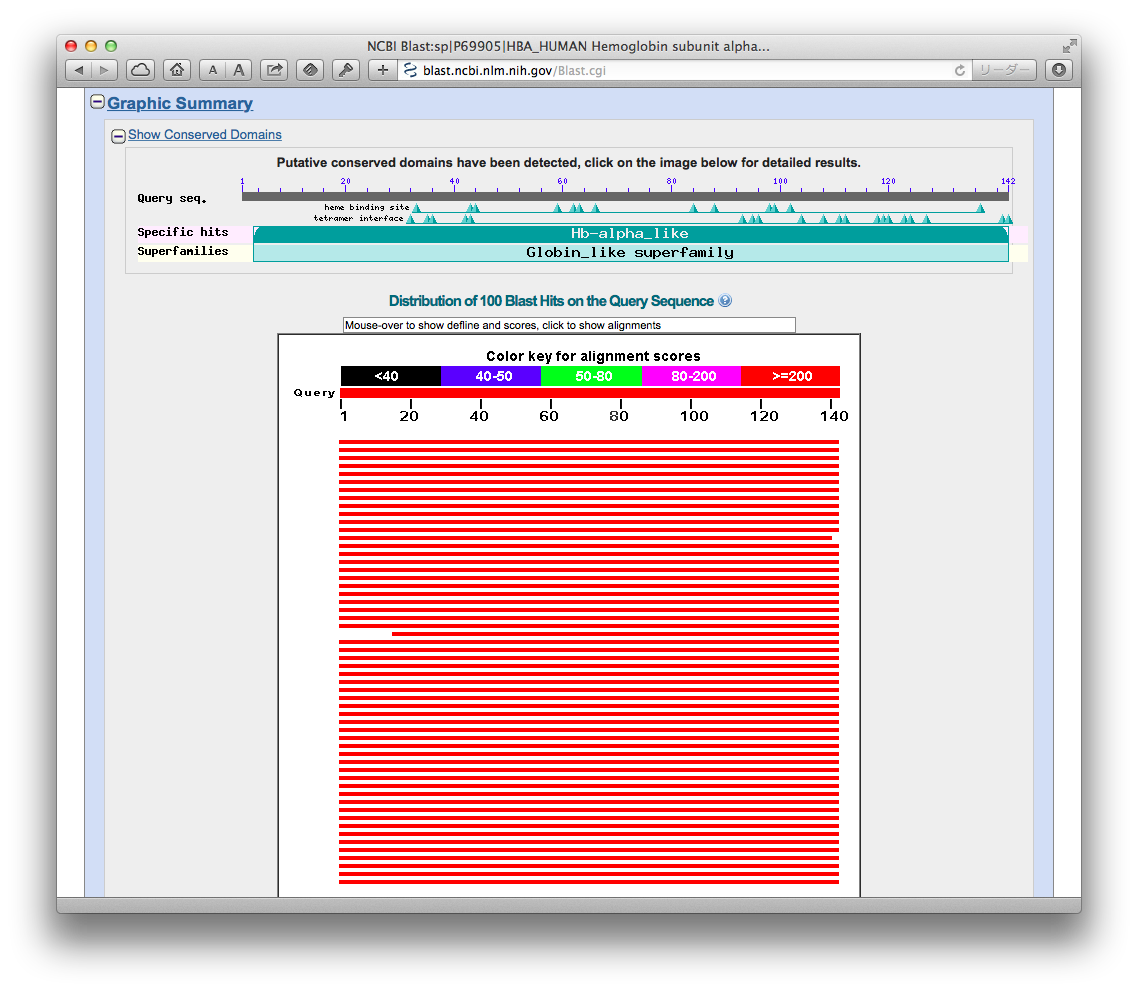
一番上には,サマリーとして,検索されたタンパク質の一致部分の一覧が図示されています.
一致部分が表示され,一致部分の長さが色で表されています.
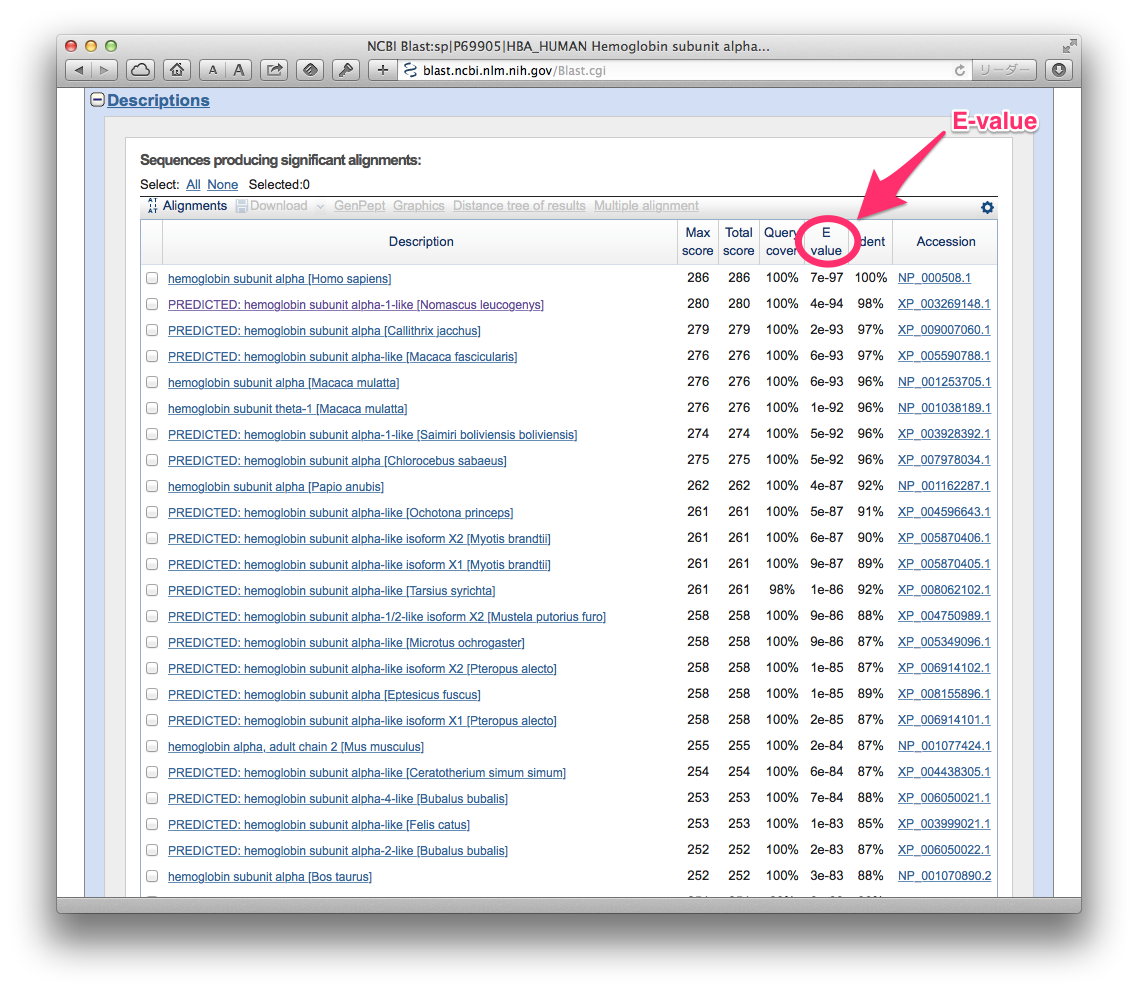
次に,検索されたタンパク質の名前,最大スコア,総合スコア,クエリーのカバー率,E-vlaue,一致率の一覧表が表示されています.
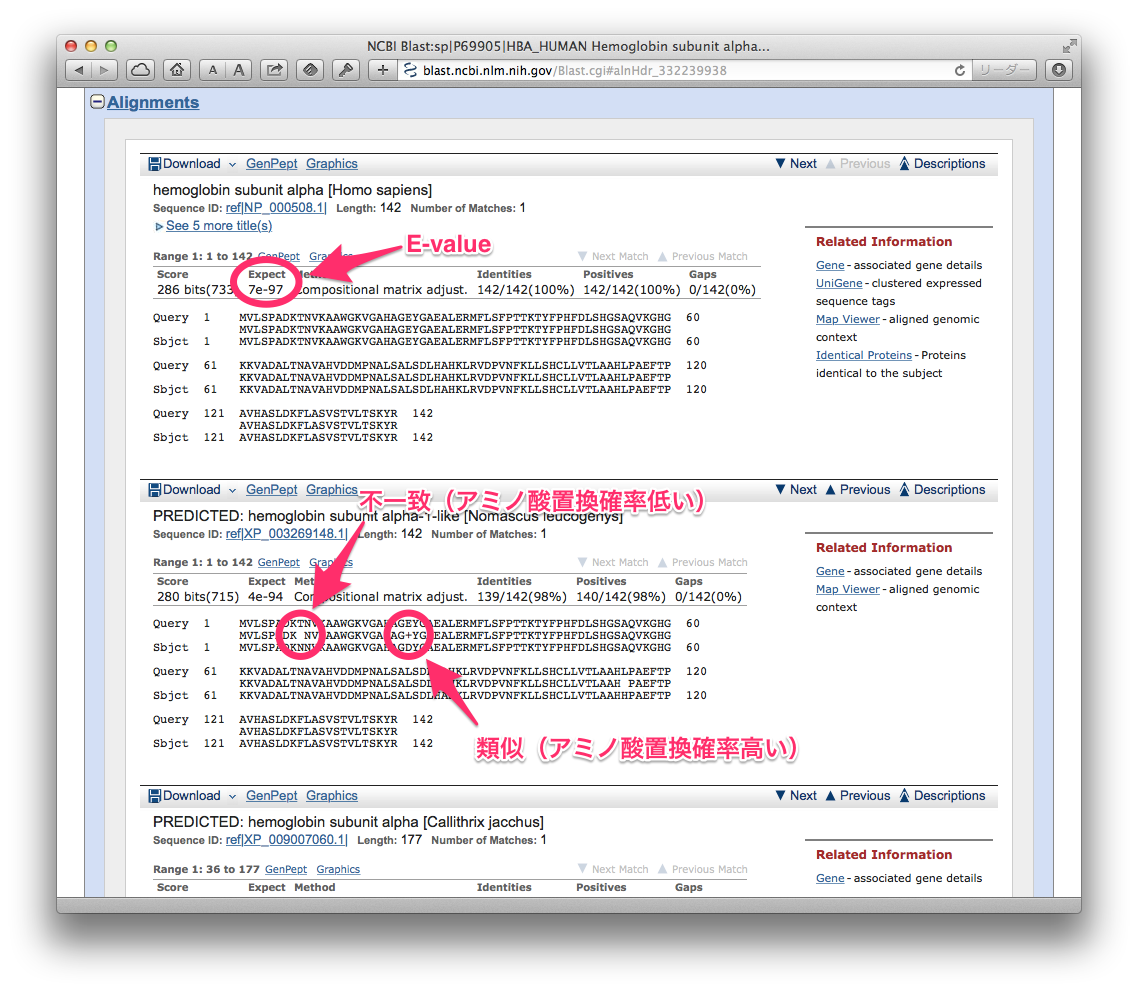
最後に,検索されたタンパク質のそれぞれについて,ペアワイズ・アラインメントの結果が表示されています.
ClustalWのアラインメント結果とは異なり,クエリー配列と検索された配列の間に一致したアミノ酸が表示されています. アミノ酸置換確率が高く,類似しているアミノ酸の所には+が表示されています.
演習 †
UniProtからヒトのアミラーゼ AMY1_HUMAN のアミノ酸配列を入手し,BLASTを用いて相同性検索を行ってみよう.
ただし,アミラーゼは配列長が長いので,丸ごと検索すると,E-valueが0になってしまいます.
一部分だけを取り出して(一部分を削除して)検索してみましょう. アミノ酸配列がどのくらいの長さだとE-valueがどのくらいになるでしょうか.
参考文献 †
- 6章 類似配列のデータベース検索
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)