- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- バイオ・データ・マイニング/Rでスペクトラル・クラスタリングを使う へ行く。
はじめに †
『Rによるバイオインフォマティクスデータ解析』の7.10節「スペクトラル・クラスタリング」を参考にして,スペクトラル・クラスタリングを行います.
準備 †
Rのインストールについては,次のページを見てください.
[math]k[/math]平均法が苦手なデータとして,二重円のデータを作成します. [math]-4[/math]から[math]4[/math]までの二次元の一様乱数を1,000個生成し,半径[math]3[/math]から[math]4[/math]の範囲にあるデータを[math]c_2[/math],半径[math]1[/math]から[math]2[/math]の範囲にあるデータを[math]c_1[/math]とします.
x = runif(1000) * 8 - 4 y = runif(1000) * 8 - 4 data = cbind(x, y) c2 = (x^2 + y^2) < 16 & (x^2 + y^2) > 9 c1 = (x^2 + y^2) < 4 & (x^2 + y^2) > 1 logo = data[(c2 | c1),] plot(logo)
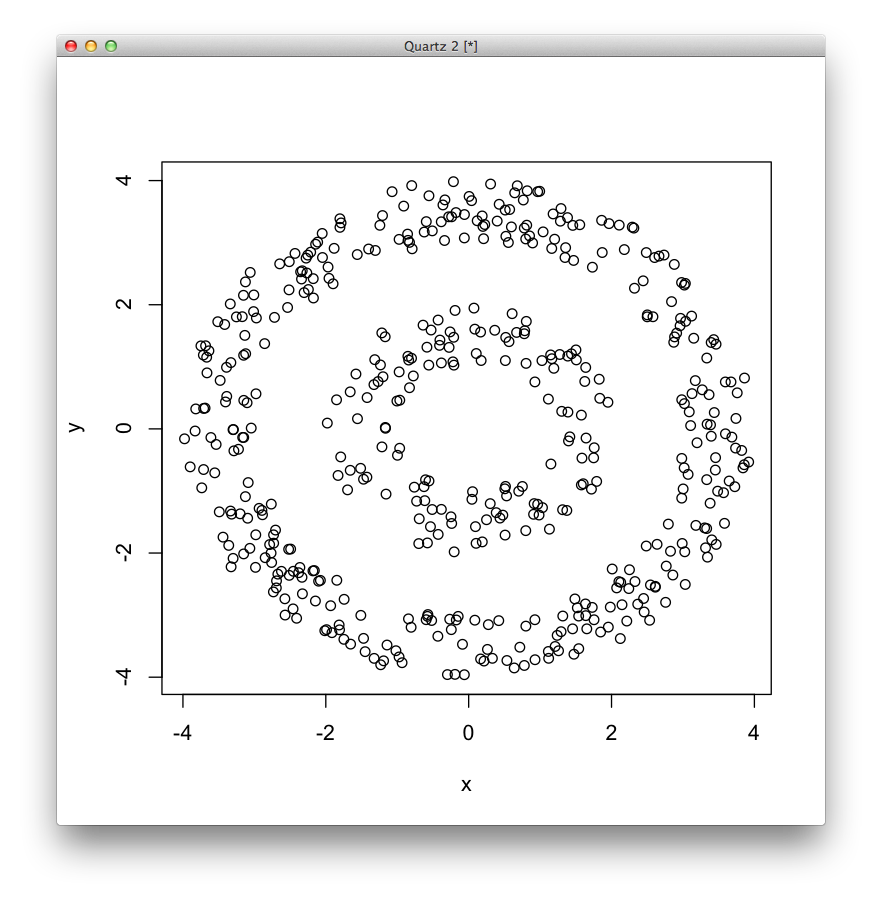
まずは[math]k[/math]平均法を試して見ます.
model = kmeans(logo, 2) plot(logo, col=model$cluster, pch=model$cluster)
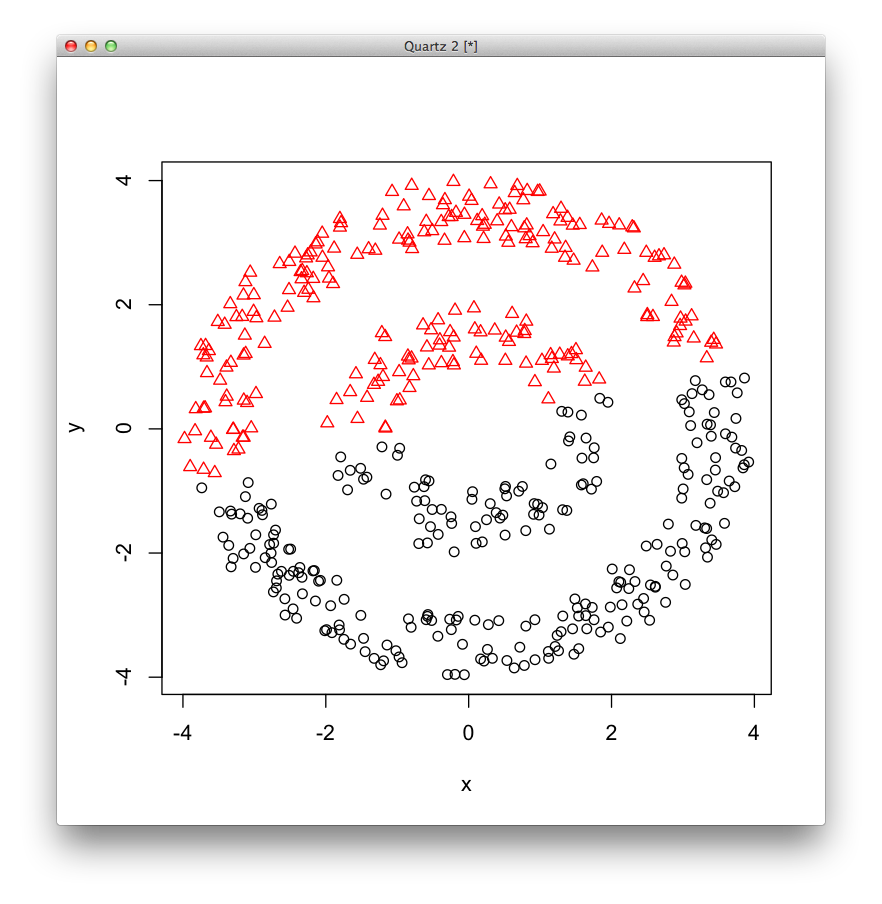
やはり[math]k[/math]平均法ではうまくクラスタリングできません.
スペクトラル・クラスタリングは,kernlabパッケージで提供されています. そこで,kernlabパッケージをインストールします.install.packagesコマンドを実行すると,パッケージをダウンロードするサーバーを聞かれますので,リストからJapanのどれかを選択します.
install.packages("kernlab") library(kernlab)
スペクトラル・クラスタリング †
スペクトラル・クラスタリングは,距離行列(類似度行列)を次元削減してそこで[math]k[/math]平均法などのクラスタリングを行う手法です.
スペクトラル・クラスタリングを行うには,speccコマンドを用います. speccコマンドには,データとクラスター数を引数として与えます.
model = specc(logo, center=2) plot(logo, col=model, pch=model)
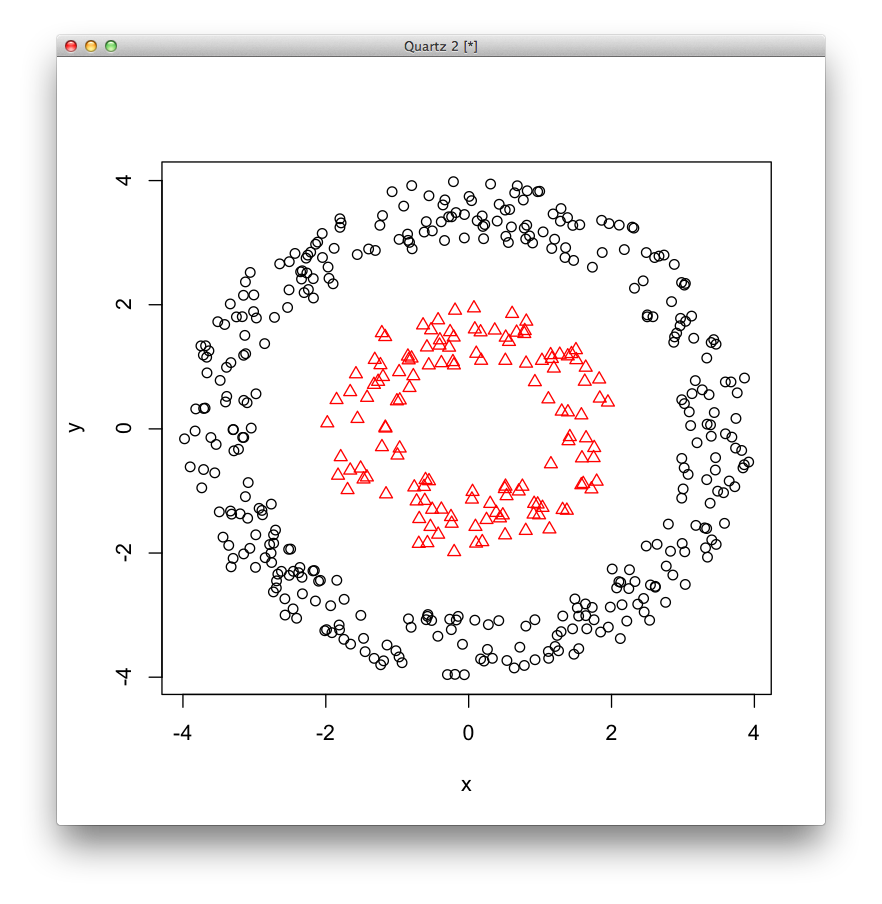
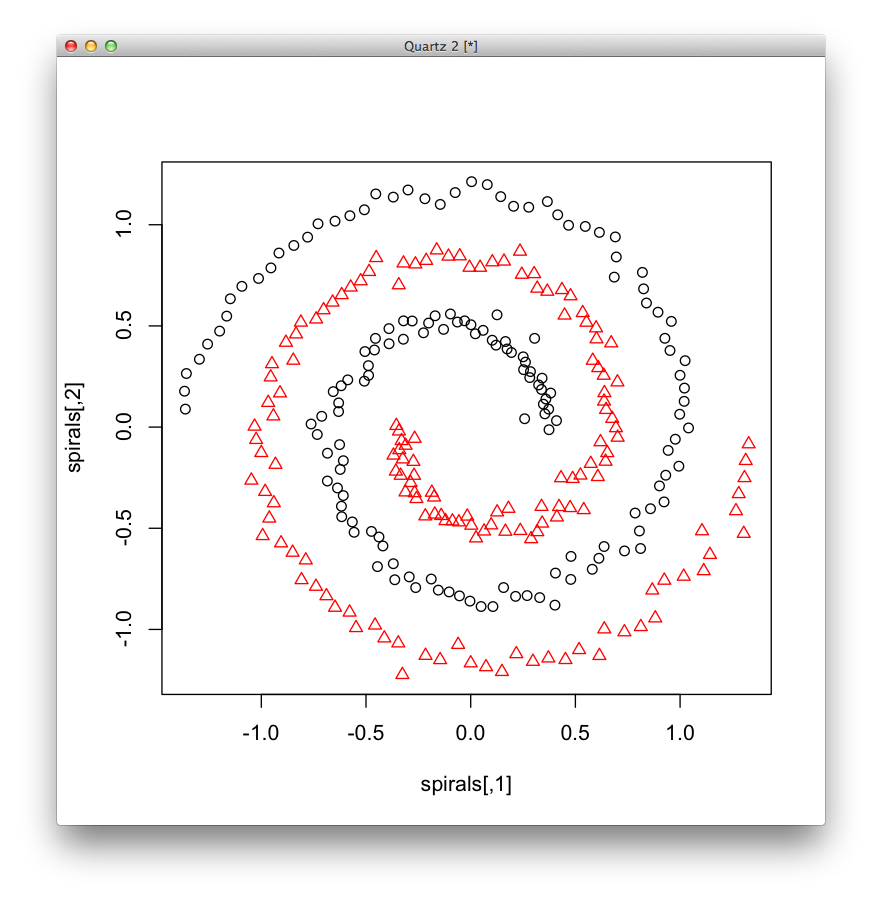
スペクトラル・クラスタリングではきれいにクラスタリングできました.
サンプル・データのspiralsでも試してみます.
data(spirals) plot(spirals)
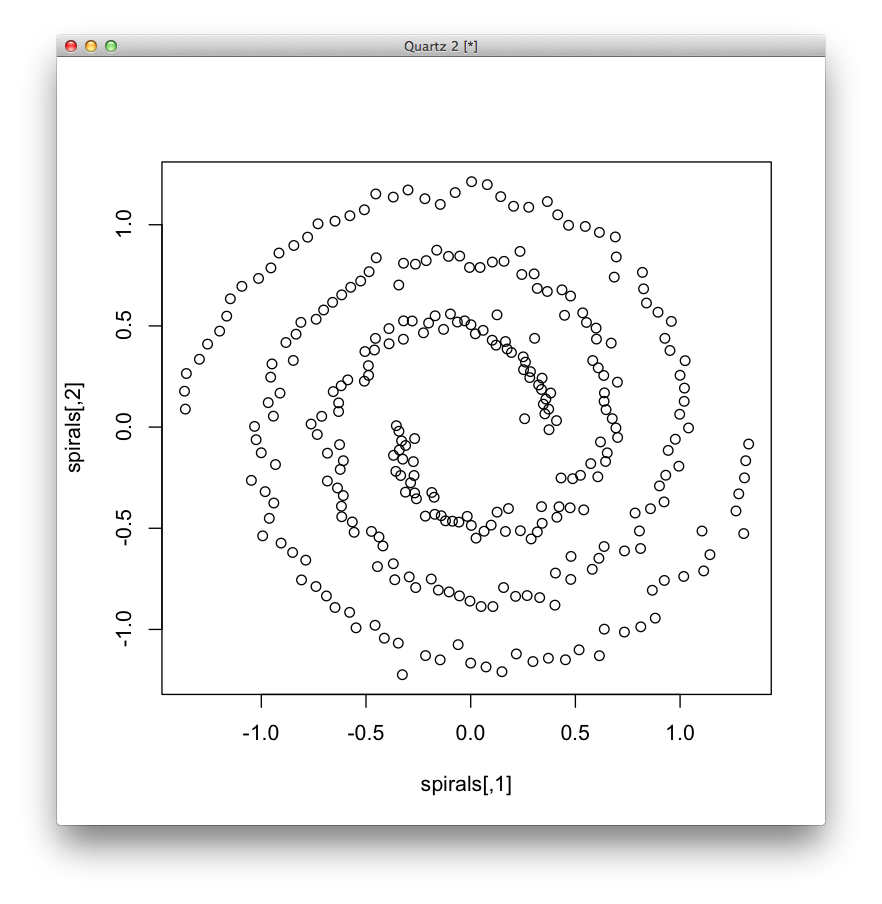
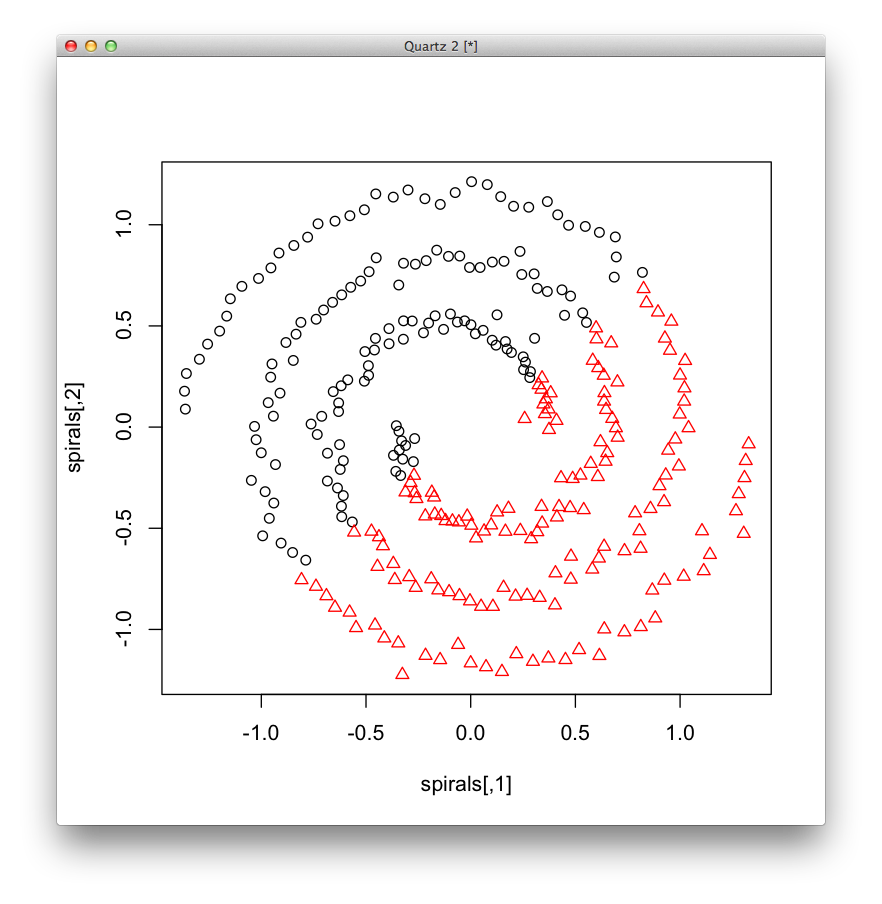
まず,[math]k[/math]平均法を試します.
model = kmeans(spirals, 2) plot(spirals, col=model$cluster, pch=model$cluster)
次に,スペクトラル・クラスタリングを用います.
model = specc(spirals, center=2) plot(spirals, col=model, pch=model)
speccは内部でランダムな初期化を行っていますので,[math]k[/math]平均法と同様に,乱数のシードによって結果が変わります. 同じ結果を得るためには,seed.setを用いて乱数のシードを指定してからスペクトラル・クラスタリングを行います.
set.seed(0) model = specc(spirals, center=2)
irisデータセット †
irisデータセットでもやってみましょう.
data(iris)
このデータセットは,アヤメの種類(Species)を花びらの長さ(Sepal.Length),幅(Lepal.Width),がくの長さ(Petal.Length),幅(Petal.Width)によって分類する問題です. 長さと幅は連続値,種類はsetosa, versicolor, virginicaのいずれかをとる離散値です.
このデータセットには,setosa, versicolor, virginicaという3種類のアヤメについて,それぞれ50個ずつ,合計150個のデータが含まれています. ランダムに10個のデータを選択して,見てみましょう.
iris[sort(sample(1:150,10)),]
ここでは,Speciesを除いた1列目から4列目だけを取り出して使います.
> iris[sort(sample(1:150,10)),] Sepal.Length Sepal.Width Petal.Length Petal.Width Species 4 4.6 3.1 1.5 0.2 setosa 22 5.1 3.7 1.5 0.4 setosa 65 5.6 2.9 3.6 1.3 versicolor 97 5.7 2.9 4.2 1.3 versicolor 100 5.7 2.8 4.1 1.3 versicolor 108 7.3 2.9 6.3 1.8 virginica 116 6.4 3.2 5.3 2.3 virginica 122 5.6 2.8 4.9 2.0 virginica 136 7.7 3.0 6.1 2.3 virginica 146 6.7 3.0 5.2 2.3 virginica
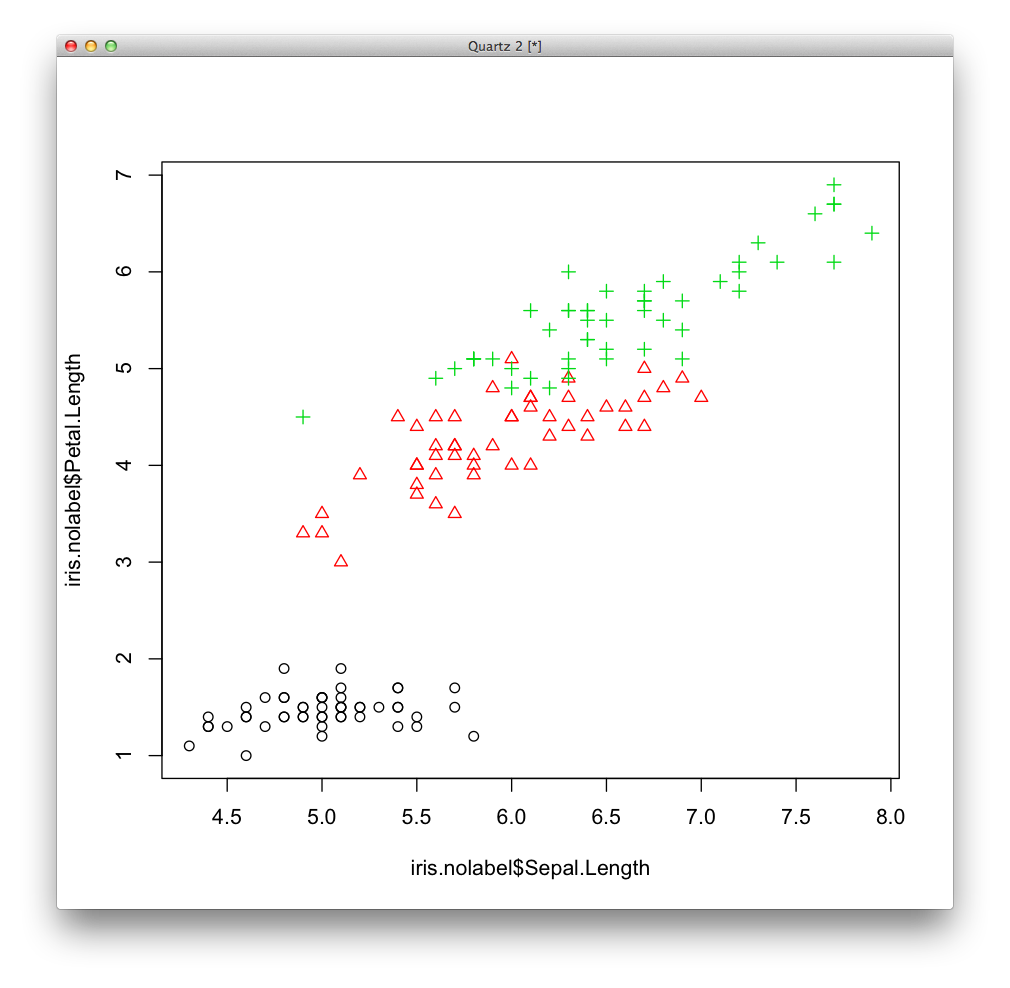
4次元データですが,このうちのSepal.LengthとPetal.Lengthだけを取り出して表示します. データを表示するときの色colと形pchにはirisのSpeciesをas.numeric関数で数値化したものを指定します.
x <- iris[1:4]
カテゴリーの数は3だとわかっていますので,クラスター数を[math]3[/math]としてスペクトラル・クラスタリングを行います.
plot(x$Sepal.Length, x$Petal.Length, col=as.numeric(iris$Species), pch=as.numeric(iris$Species))
今度は色と形をクラスタリングによって得られたクラスター番号によって指定して表示します.
model <- specc(x, center=3)
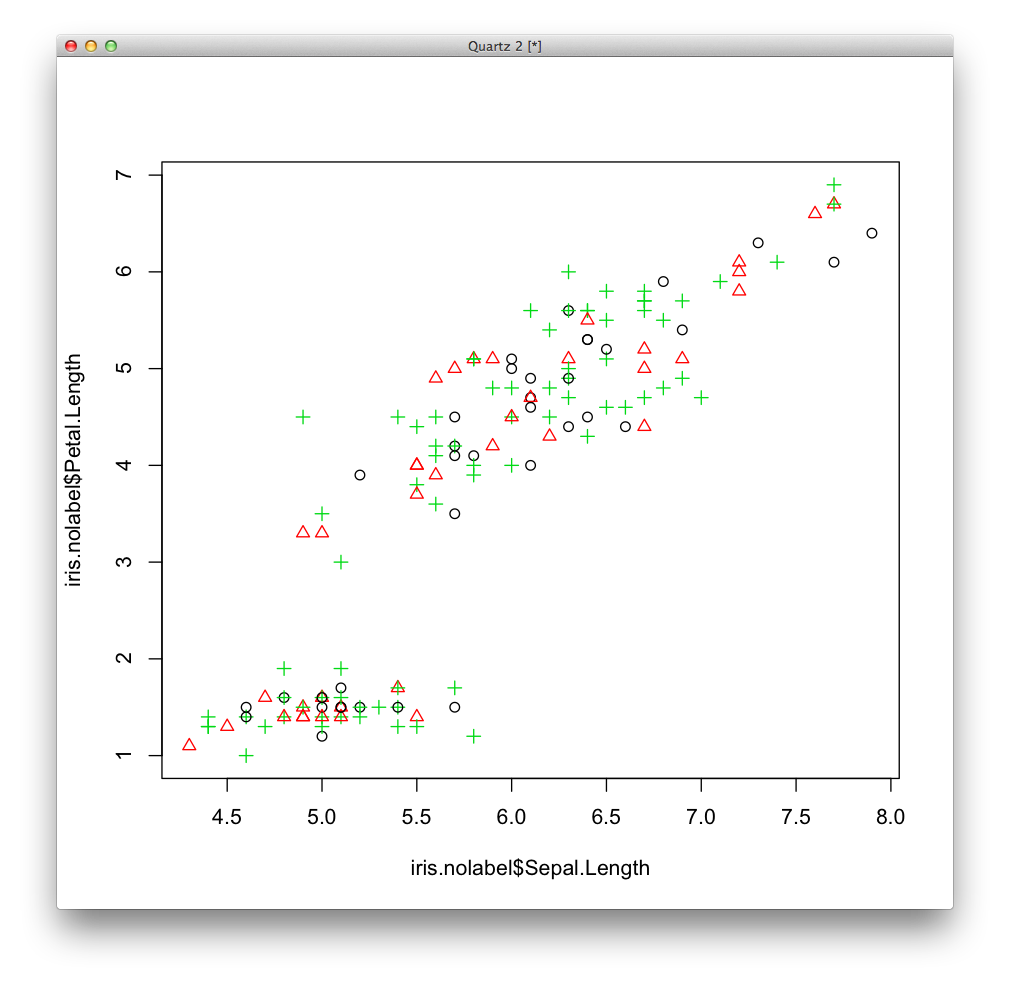
残念ながら,irisデータセットはスペクトラル・クラスタリングではうまくクラスタリングできませんでした.
まとめ †
スペクトラル・クラスタリングは,距離行列(類似度行列)を次元削減してそこで[math]k[/math]平均法などのクラスタリングを行う手法です.
[math]k[/math]平均法が苦手とするようなデータでもうまくいくことがあります.
(内部で行われているクラスタリングが乱数に依存する手法の場合)スペクトラル・クラスタリングの結果は乱数のシードに依存しますので,乱数のシードを変えて実行するとよりよい結果が得られる可能性があります.
参考文献 †
- クラスタリングとは(クラスター分析とは) - 神嶌敏弘さん
- スペクトラルクラスタリングは次元圧縮しながらKmeansする手法 - 観月橋日記 (続生駒日記)
- Rでk平均法を使う - とうごろうぃき
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)