- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- バイオ・データ・マイニング/Rでスペクトラル・クラスタリングを使う へ行く。
はじめに †
『Rによるバイオインフォマティクスデータ解析』の7.10節「スペクトラル・クラスタリング」を参考にして,スペクトラル・クラスタリングを行います.
準備 †
Rのインストールについては,次のページを見てください.
[math]k[/math]平均法が苦手なデータとして,二重円のデータを作成します. [math]-4[/math]から[math]4[/math]までの二次元の一様乱数を1,000個生成し,半径[math]3[/math]から[math]4[/math]の範囲にあるデータを[math]c_2[/math],半径[math]1[/math]から[math]2[/math]の範囲にあるデータを[math]c_1[/math]とします.
x = runif(1000) * 8 - 4 y = runif(1000) * 8 - 4 data = cbind(x, y) c2 = (x^2 + y^2) < 16 & (x^2 + y^2) > 9 c1 = (x^2 + y^2) < 4 & (x^2 + y^2) > 1 logo = data[(c2 | c1),] plot(logo)
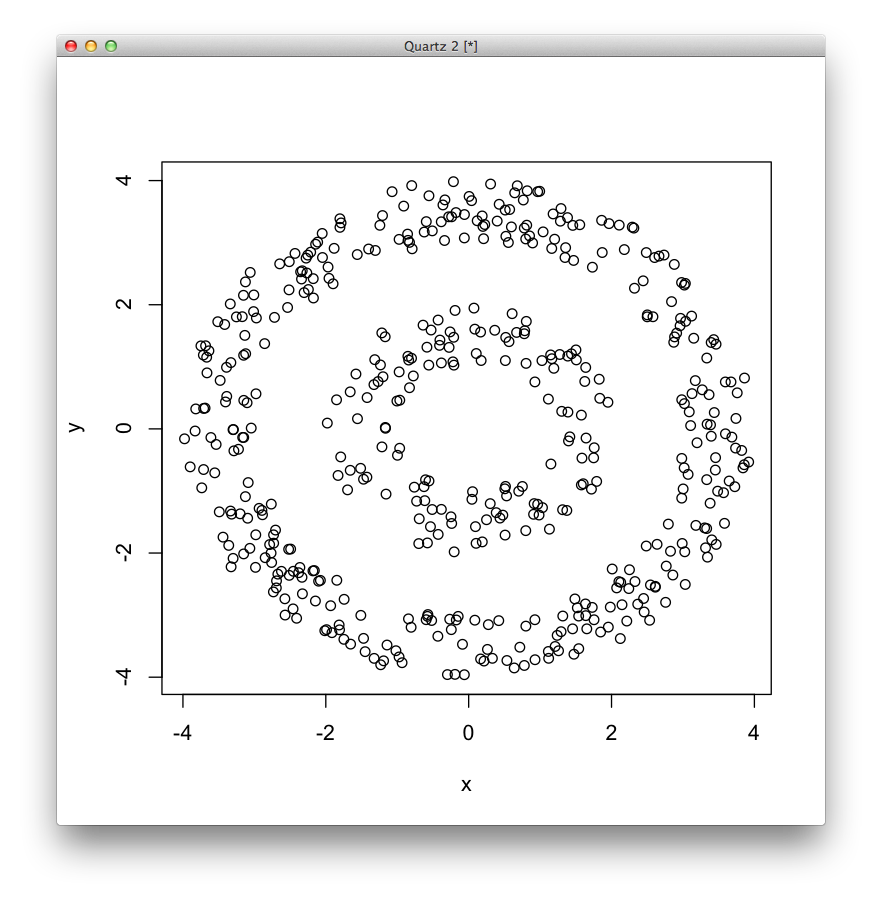
まずは[math]k[/math]平均法を試して見ます.
model = kmeans(logo, 2) plot(logo, col=model$cluster, pch=model$cluster)
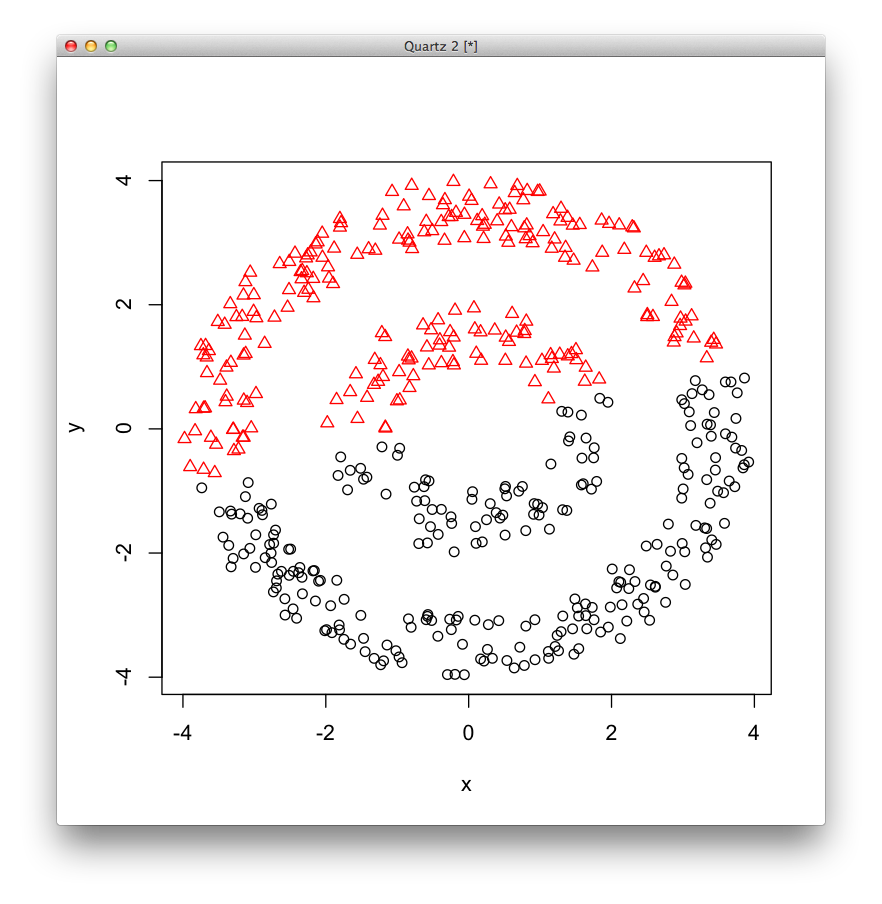
やはり[math]k[/math]平均法ではうまくクラスタリングできません.
スペクトラル・クラスタリングは,kernlabパッケージで提供されています. そこで,kernlabパッケージをインストールします.install.packagesコマンドを実行すると,パッケージをダウンロードするサーバーを聞かれますので,リストからJapanのどれかを選択します.
install.packages("kernlab") library(kernlab)
スペクトラル・クラスタリング †
スペクトラル・クラスタリングは,距離行列(類似度行列)を次元削減してそこで[math]k[/math]平均法などのクラスタリングを行う手法です.
スペクトラル・クラスタリングを行うには,speccコマンドを用います. speccコマンドには,データとクラスター数を引数として与えます.
model = specc(logo, center=2) plot(logo, col=model, pch=model)
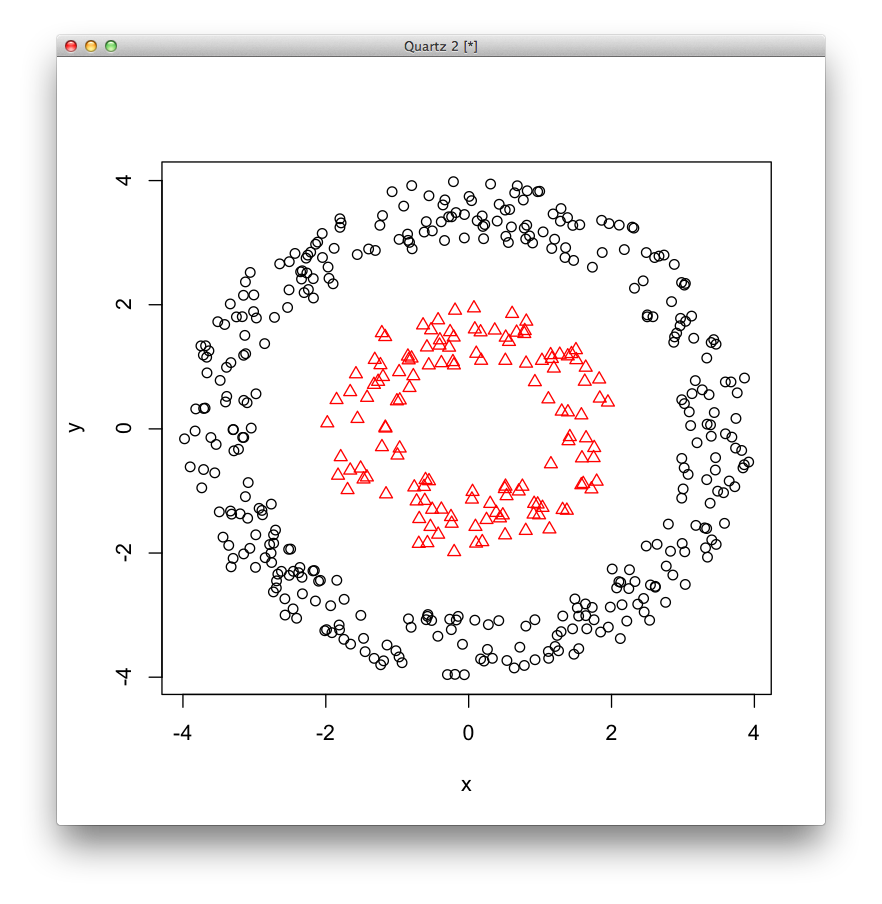
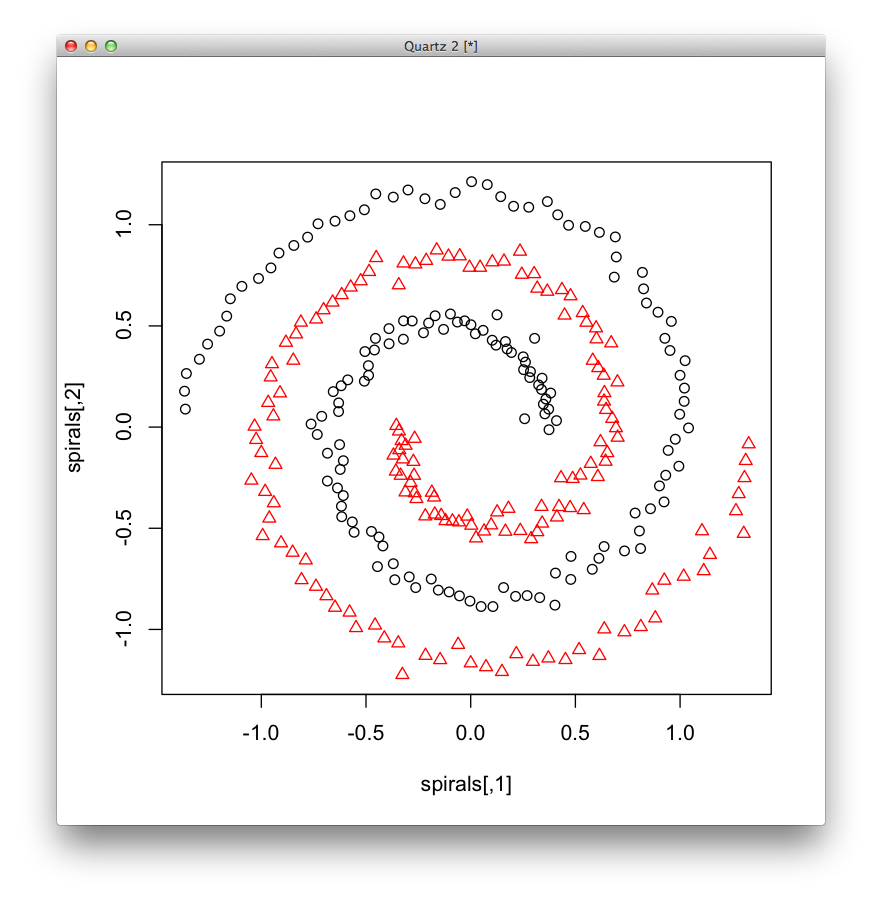
スペクトラル・クラスタリングではきれいにクラスタリングできました.
サンプル・データのspiralsでも試してみます.
data(spirals) plot(spirals)
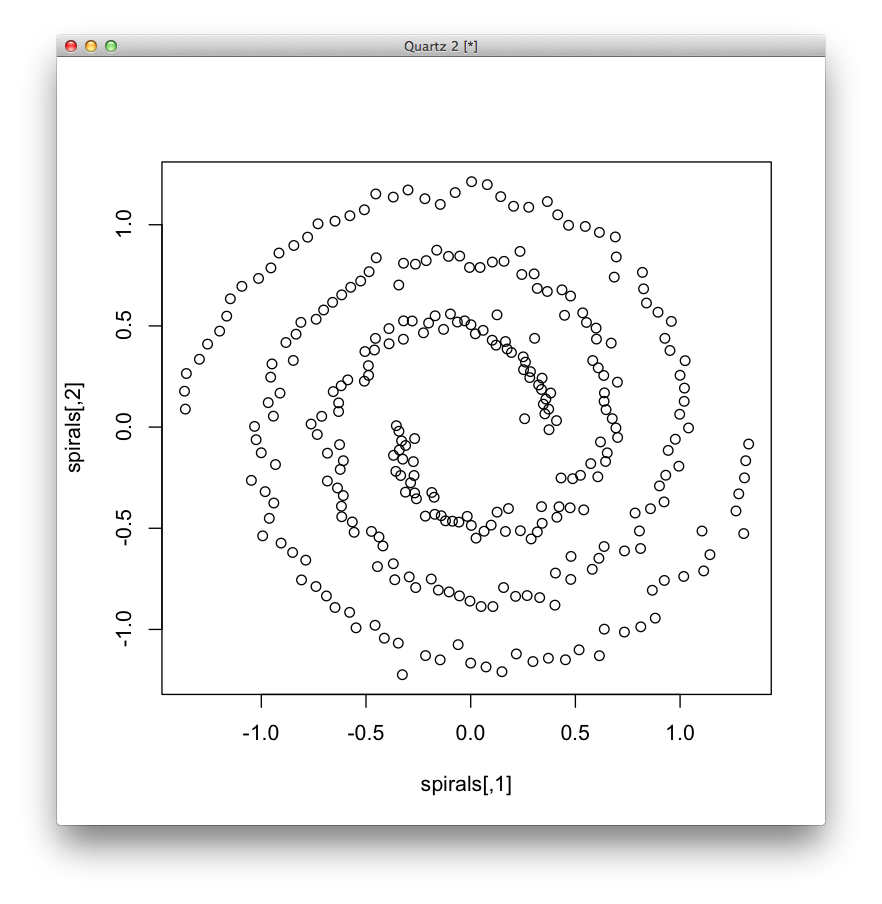
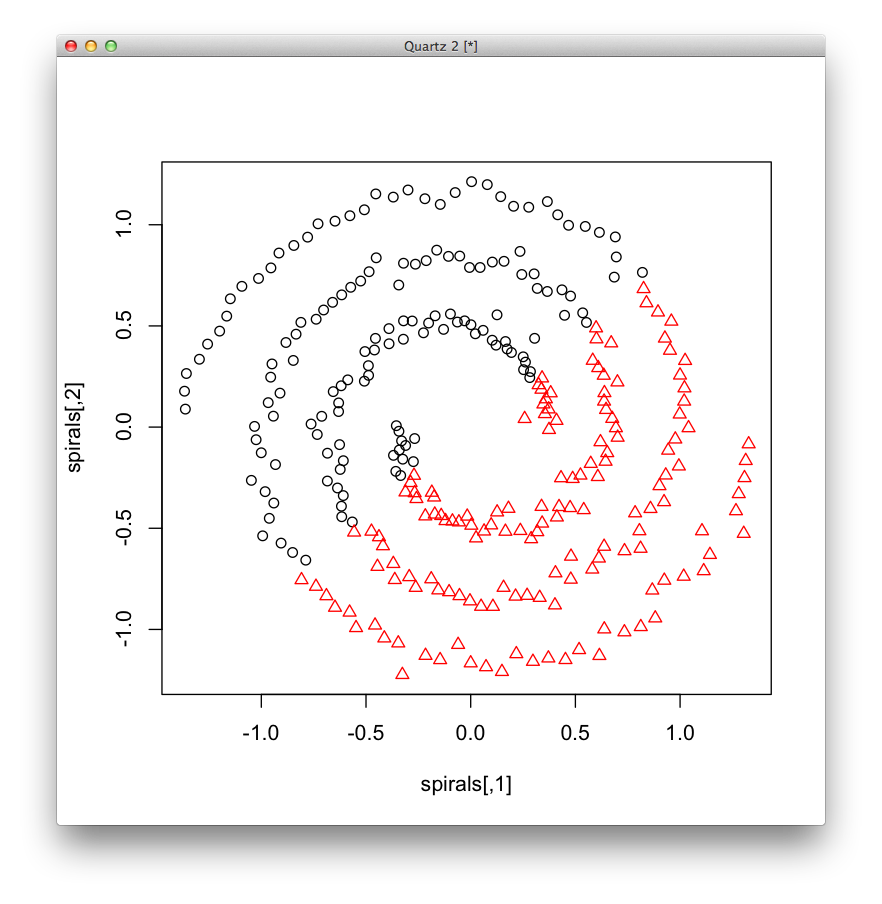
まず,[math]k[/math]平均法を試します.
model = kmeans(spirals, 2) plot(spirals, col=model$cluster, pch=model$cluster)
次に,スペクトラル・クラスタリングを用います.
model = specc(spirals, center=2) plot(spirals, col=model, pch=model)
speccは内部でランダムな初期化を行っていますので,[math]k[/math]平均法と同様に,乱数のシードによって結果が変わります. 同じ結果を得るためには,seed.setを用いて乱数のシードを指定してからスペクトラル・クラスタリングを行います.
set.seed(0) model = specc(spirals, center=2)
まとめ †
スペクトラル・クラスタリングは,距離行列(類似度行列)を次元削減してそこで[math]k[/math]平均法などのクラスタリングを行う手法です.
[math]k[/math]平均法が苦手とするようなデータでもうまくいくことがあります.
(内部で行われているクラスタリングが乱数に依存する手法の場合)スペクトラル・クラスタリングの結果は乱数のシードに依存しますので,乱数のシードを変えて実行するとよりよい結果が得られる可能性があります.
参考文献 †
- クラスタリングとは(クラスター分析とは) - 神嶌敏弘さん
- スペクトラルクラスタリングは次元圧縮しながらKmeansする手法 - 観月橋日記 (続生駒日記)
- Rでk平均法を使う - とうごろうぃき
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)