- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- バイオ・データ・マイニング/Rでk平均法を使う へ行く。
この記事はまだ書きかけです.
はじめに †
『Rによるバイオインフォマティクスデータ解析』の7.9.1節「k-menas」を参考にして,k平均法を行います.
準備 †
Rのインストールについては,次のページを見てください.
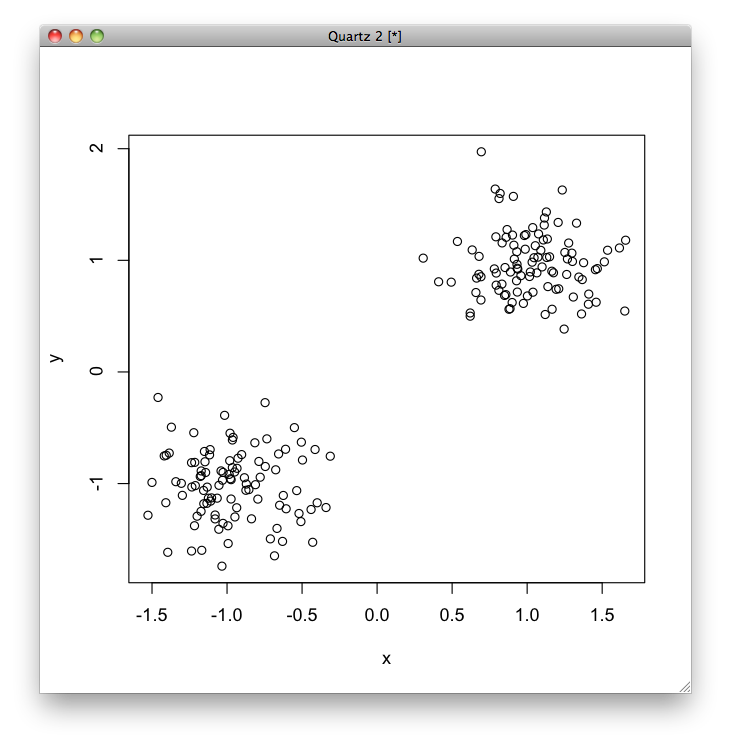
まず,[math](1, 1)[/math] を中心として,[math]x[/math]座標と[math]y[/math]座標をそれぞれ分散0.3として正規分布で100個の点を生成し,これをc1とします. 次に,[math](-1 -1)[/math] を中心として,同じように分散0.3の正規分布で100個の点を生成し,これをc2とします. c1とc2をまとめて,dataとし,これをプロットします.
set.seed(123) x1 = rnorm(100, mean=1, sd=0.3) y1 = rnorm(100, mean=1, sd=0.3) c1 <- cbind(x1, y1) x2 = rnorm(100, mean=-1, sd=0.3) y2 = rnorm(100, mean=-1, sd=0.3) c2 <- cbind(x2, y2) data1 <- rbind(c1, c2) colnames(data1) <- c("x", "y") plot(data1)
クラスタリング †
クラスタリングは,分類対象のデータ集合をいくつかのグループに分割するものです. 分割された部分データ集合をクラスターといいます.
クラスタリングの手法には,主に階層的アプローチと分割最適化アプローチがあります. ここでは,後者の分割最適化アプローチの一つであるk平均法を行います.
k平均法 †
[math]k[/math]平均法([math]k[/math]-means)は,データの集合を [math]k[/math] 個のクラスターに分割します. クラスター数 [math]k[/math] を最初に決めておかなければなりません.
[math]k[/math]平均法は,次の手順で行います.
- 各データ [math]\bf{x}_1, \dots, \bf{x}_n[/math] に対して,ランダムにクラスター [math]c_1, \dots, c_k[/math] を割り当てる.
- クラスター [math]c_1, \dots, c_k[/math] ごとに,クラスター [math]c_i[/math] に割り当てられたデータの平均を求め,それを [math]c_i[/math] の中心とする.
- 各データ [math]\bf{x}_1, \dots, \bf{x}_n[/math] に対して,データ [math]\bf{x}_i[/math] と各クラスター [math]c_1, \dots, c_k[/math] の中心との距離を求め,[math]\bf{x}_i[/math] を最も中心が近いクラスターに割り当て直す.
- 全てのデータについてクラスターの割り当てが変更されなかったら終了する.そうでない場合は,ステップ2へ戻る.
Rでk平均法を用いるには,kmeans関数を用います. kmeans関数の引数には,分割するデータとクラスター数 [math]k[/math] を与えます.
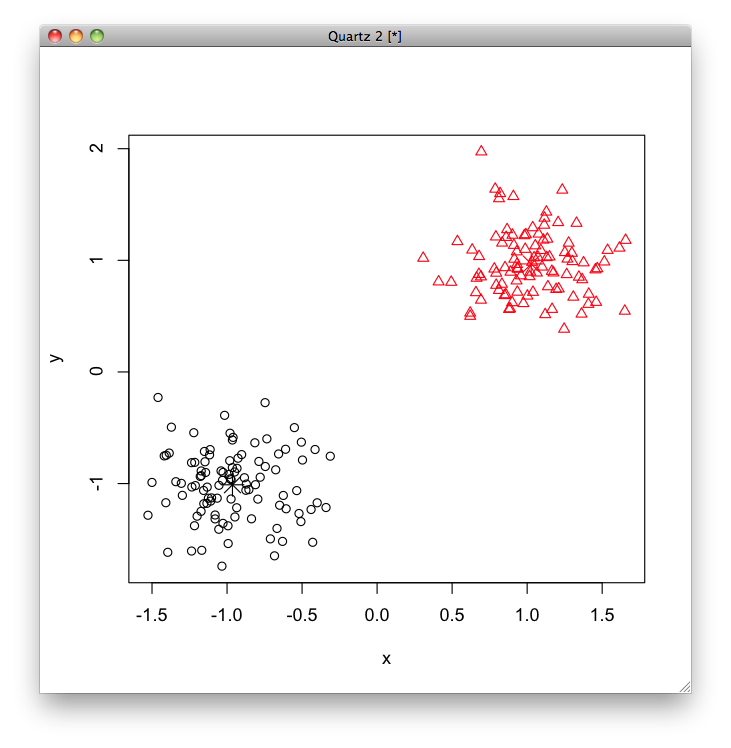
model <- kmeans(data1, 2)
作成されたオブジェクトのcluster変数には割り当てられたクラスター番号が,center変数には各クラスターの中心座標が格納されます.
model$cluster
結果をプロットすると次のようになります.
> model$cluster [1] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 [26] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 [51] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 [76] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 [101] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 [126] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 [151] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 [176] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
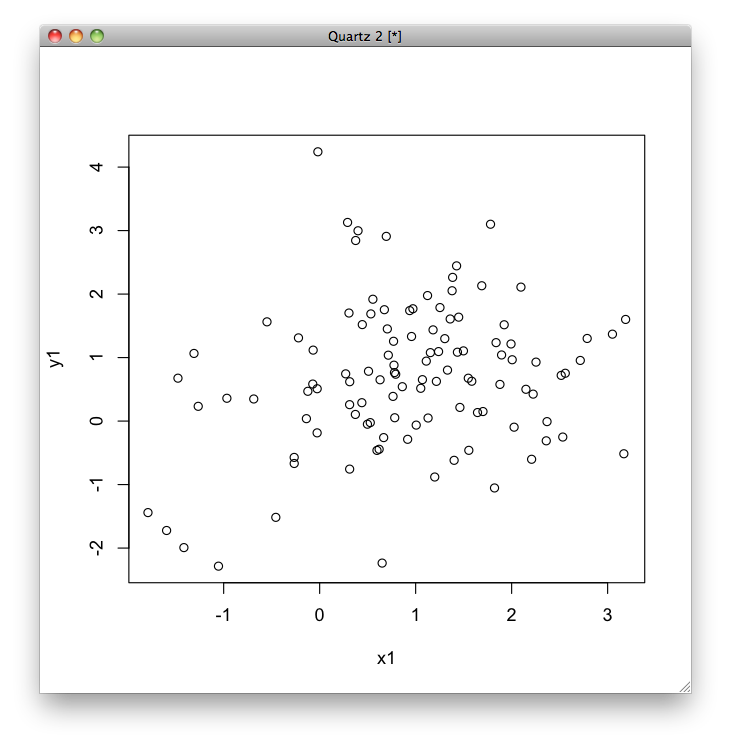
今度は,もう少し分散を大きくし,かつ,データの個数を同じにしないでやってみます.
model$center
> model$center x y 1 -0.9638605 -1.010867 2 1.0271218 0.967736
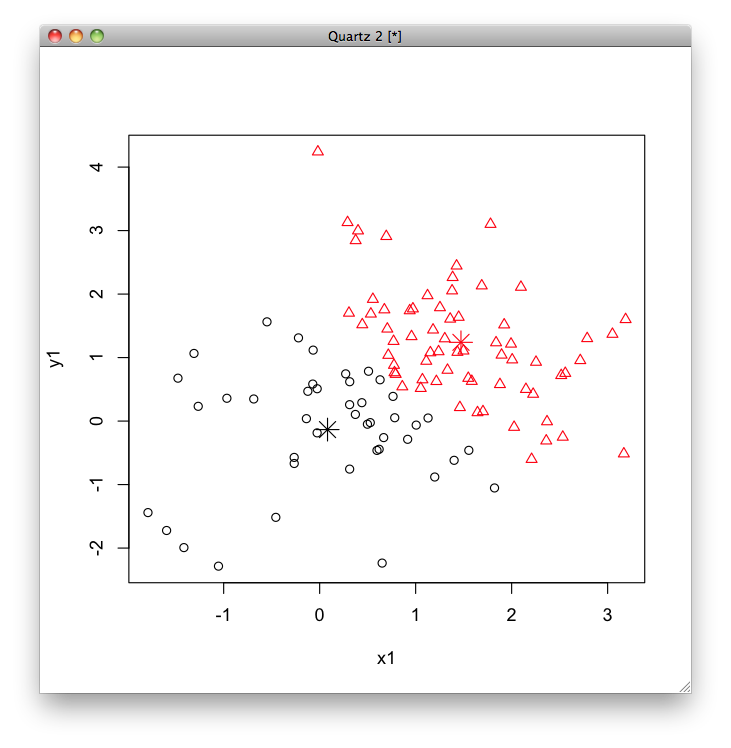
すると,データを生成するときに使用した中心 [math](1, 1), (-1, -1)[/math] よりもずれてしまいました. こういうデータは苦手です.
まとめ †
この記事はまだ書きかけです.
参考文献 †
- クラスタリングとは(クラスター分析とは) - 神嶌敏弘さん
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)