- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- バイオ・データ・マイニング/ClustalWで多重アラインメントを行う へ行く。
- 1 (2014-10-03 (金) 11:28:13)
- 2 (2014-10-03 (金) 19:08:13)
- 3 (2014-10-09 (木) 03:57:24)
- 4 (2014-10-09 (木) 09:49:48)
- 5 (2014-10-10 (金) 07:12:22)
- 6 (2014-10-10 (金) 07:15:59)
- 7 (2014-10-10 (金) 10:53:12)
- 8 (2014-10-17 (金) 07:36:57)
- 9 (2014-10-17 (金) 07:36:57)
- 10 (2019-10-09 (水) 13:15:57)
- 11 (2024-03-22 (金) 11:57:40)
- 12 (2025-09-12 (金) 19:37:09)
はじめに †
ここでは、ClustalWを使ってタンパク質のアミノ酸配列の多重アラインメントを行います。
ClustalWの基本的な使い方については、以下のページを参照してください。
- ClustalWでペアワイズ・アラインメントを行う - とうごろうぃき
ここでは、系統樹を表示できるGenomeNetのClustalWを使います。
多重アラインメント †
3本以上の配列に対する配列アラインメント(シーケンス・アラインメント)を多重配列アラインメント(多重アラインメント、マルチアラインメント)と言います。
多重アラインメントの仕組み †
ここではClustalWによる多重アラインメントについて説明します。 その他の方法もありますが、ここでは説明しません。
ClustalWは、累進法と呼ばれる方法を用いて多重アラインメントを行います。
まずはじめに、すべての配列からペアを作成し、ペアワイズ・アラインメントを行ってスコア(距離行列)を求めます。
次に、このスコア(距離行列)に基づいて、近隣結合法(NJ)を用いて階層クラスタリングを行い、案内木(guide tree)と呼ばれるデンドログラムを作成します。
最後に、案内木に従って、多重アラインメントを作成します。
近隣結合法 (NJ) †
近隣結合法は、枝の長さが細小になるような近隣クラスターを結合することを繰り返す方法です。 この方法では、根がない木(無根系統樹)を作成します。
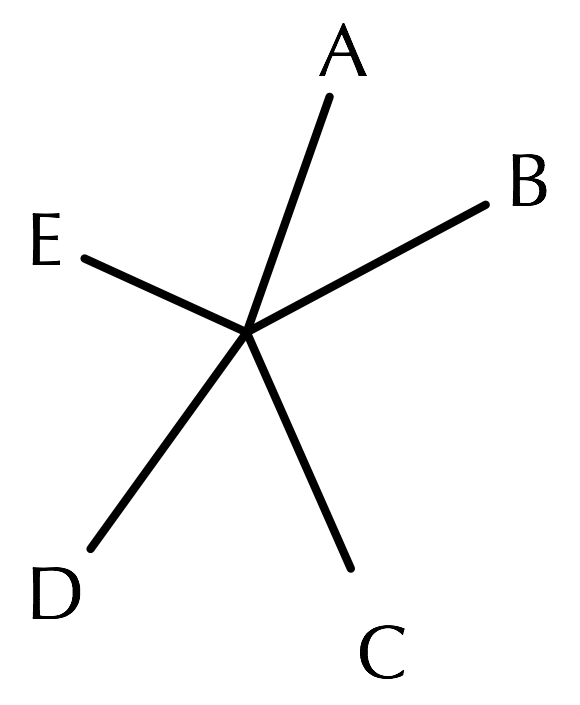
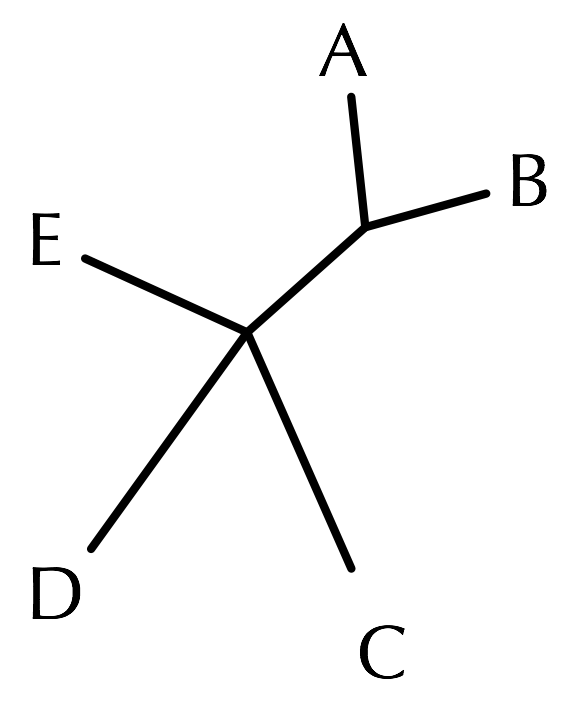
A, B, C, D, E という5つの種があり、ペアワイズ・アラインメントによって距離を求めたところ A と B の間の距離が最も短いとします。
このとき、近隣結合法では、まずはじめにそれぞれの種を操作上の分類ユニットOTU (Operational Taxonomic Unit)とし、全てのOTUに連結した節を作ります。
それから、最も近いOTUである A と B を結合してノードを作り、このノードと葉 A, B を含む部分木を新しいOTUとし、新しい枝を作ります。
これを繰り返します。
genome.jpのCLUSTALWでは、NJの改良版であるBIONJとfasttreeを使うことができます。
タンパク質の多重アラインメント †
タンパク質データベースの一つであるUniProtからタンパク質のアミノ酸配列を取得して、ClustalWで多重アラインメントを行います。
UniProtからアミノ酸配列を取得する方法については、以下のページを参照してください。
- ClustalWでペアワイズ・アラインメントを行う - とうごろうぃき
ここでは、以下のヘモグロビン(Hemoglobin subunit alpha)のアミノ酸配列に対してペアワイズ・アラインメントをやってみます。
- HBA_HUMAN ヒト
- HBA_RAT ラット
- HBA_MOUSE マウス
- HBA_BOVIN 牛
- HBA_RABIT ウサギ
- HBA_CHICK 鶏
- HBA_HORSE 馬
- HBA_PANTR チンパンジー
- HBA_DANRE ゼブラフィッシュ
- HBA_EQUHE ロバ
- HBA_XENTR カエル
- HBA_PANPA ボノボ
- HBA_CYPCA 鯉
- HBA_LEPWE アザラシ
- HBA_SHEEP 羊
まず、UniProtで Hemoglobin HBA を検索し、アミノ酸配列を入手します。
次に、ClustalWにかけると、次のような多重アラインメントが得られます。
CLUSTAL 2.1 multiple sequence alignment
HBA_RAT MVLSADDKTNIKNCWGKIGGHGGEYGEEALQRMFAAFPTTKTYFSHI-DVSPGSAQVKAH
HBA_MOUSE MVLSGEDKSNIKAAWGKIGGHGAEYGAEALERMFASFPTTKTYFPHF-DVSHGSAQVKGH
HBA_HUMAN MVLSPADKTNVKAAWGKVGAHAGEYGAEALERMFLSFPTTKTYFPHF-DLSHGSAQVKGH
HBA_PANTR MVLSPADKTNVKAAWGKVGAHAGEYGAEALERMFLSFPTTKTYFPHF-DLSHGSAQVKGH
HBA_PANPA MVLSPADKTNVKAAWGKVGAHAGEYGAEALERMFLSFPTTKTYFPHF-DLSHGSAQVKGH
HBA_BOVIN MVLSAADKGNVKAAWGKVGGHAAEYGAEALERMFLSFPTTKTYFPHF-DLSHGSAQVKGH
HBA_SHEEP MVLSAADKSNVKAAWGKVGGNAGAYGAEALERMFLSFPTTKTYFPHF-DLSHGSAQVKGH
HBA_HORSE MVLSAADKTNVKAAWSKVGGHAGEYGAEALERMFLGFPTTKTYFPHF-DLSHGSAQVKAH
HBA_EQUHE MVLSAADKTNVKAAWSKVGGNAGDFGAEALERMFLGFPTTKTYFPHF-DLSHGSAQVKAH
HBA_RABIT MVLSPADKTNIKTAWEKIGSHGGEYGAEAVERMFLGFPTTKTYFPHF-DFTHGSEQIKAH
HBA_LEPWE -VLSPADKTNVKTTWDKIGGHAGEYGGEALERTFMAFPTTKTYFPHF-DLSPGSAQVKTH
HBA_CHICK MVLSAADKNNVKGIFTKIAGHAEEYGAETLERMFTTYPPTKTYFPHF-DLSHGSAQIKGH
HBA_XENTR MHLTADDKKHIKAIWPSVAAHGDKYGGEALHRMFMCAPKTKTYFPDF-DFSEHSKHILAH
HBA_DANRE MSLSDTDKAVVKAIWAKISPKADEIGAEALARMLTVYPQTKTYFSHWADLSPGSGPVKKH
HBA_CYPCA MSLSDKDKAAVKGLWAKISPKADDIGAEALGRMLTVYPQTKTYFAHWADLSPGSGPVKKH
*: ** :* : .:. :. * *:: * : * *****.. *.: * : *
HBA_RAT GKKVADALAKAADHVEDLPGALSTLSDLHAHKLRVDPVNFKFLSHCLLVTLACHHPGDFT
HBA_MOUSE GKKVADALASAAGHLDDLPGALSALSDLHAHKLRVDPVNFKLLSHCLLVTLASHHPADFT
HBA_HUMAN GKKVADALTNAVAHVDDMPNALSALSDLHAHKLRVDPVNFKLLSHCLLVTLAAHLPAEFT
HBA_PANTR GKKVADALTNAVAHVDDMPNALSALSDLHAHKLRVDPVNFKLLSHCLLVTLAAHLPAEFT
HBA_PANPA GKKVADALTNAVAHVDDMPNALSALSDLHAHKLRVDPVNFKLLSHCLLVTLAAHLPAEFT
HBA_BOVIN GAKVAAALTKAVEHLDDLPGALSELSDLHAHKLRVDPVNFKLLSHSLLVTLASHLPSDFT
HBA_SHEEP GEKVAAALTKAVGHLDDLPGTLSDLSDLHAHKLRVDPVNFKLLSHSLLVTLACHLPNDFT
HBA_HORSE GKKVGDALTLAVGHLDDLPGALSNLSDLHAHKLRVDPVNFKLLSHCLLSTLAVHLPNDFT
HBA_EQUHE GKKVGDALTLAVGHLDDLPGALSNLSDLHAHKLRVDPVNFKLLSHCLLSTLAVHLPNDFT
HBA_RABIT GKKVSEALTKAVGHLDDLPGALSTLSDLHAHKLRVDPVNFKLLSHCLLVTLANHHPSEFT
HBA_LEPWE GKKVADALTTAVSHIDDLPGALSALSDLHAYKLRVDPVNFKLLSHCLLVTLACHHPADFT
HBA_CHICK GKKVVAALIEAANHIDDIAGTLSKLSDLHAHKLRVDPVNFKLLGQCFLVVVAIHHPAALT
HBA_XENTR GKKVSDALNEACNHLDNIAGCLSKLSDLHAYDLRVDPGNFPLLAHQILVVVAIHFPKQFD
HBA_DANRE GKTIMGAVGEAISKIDDLVGGLAALSELHAFKLRVDPANFKILSHNVIVVIAMLFPADFT
HBA_CYPCA GKVIMGAVGDAVSKIDDLVGGLAALSELHAFKLRVDPANFKILAHNVIVVIGMLYPGDFP
* : *: * ::::: . *: **:***..***** ** :*.: .: .:. * :
HBA_RAT PAMHASLDKFLASVSTVLTSKYR
HBA_MOUSE PAVHASLDKFLASVSTVLTSKYR
HBA_HUMAN PAVHASLDKFLASVSTVLTSKYR
HBA_PANTR PAVHASLDKFLASVSTVLTSKYR
HBA_PANPA PAVHASLDKFLASVSTVLTSKYR
HBA_BOVIN PAVHASLDKFLANVSTVLTSKYR
HBA_SHEEP PAVHASLDKFLANVSTVLTSKYR
HBA_HORSE PAVHASLDKFLSSVSTVLTSKYR
HBA_EQUHE PAVHASLDKFLSTVSTVLTSKYR
HBA_RABIT PAVHASLDKFLANVSTVLTSKYR
HBA_LEPWE PAVHASLDKFFSAVSTVLTSKYR
HBA_CHICK PEVHASLDKFLCAVGTVLTAKYR
HBA_XENTR PATHKALDKFLVSVSNVLTSKYR
HBA_DANRE PEVHVSVDKFFNNLALALSEKYR
HBA_CYPCA PEVHMSVDKFFQNLALALSEKYR
* * ::***: :. .*: ***
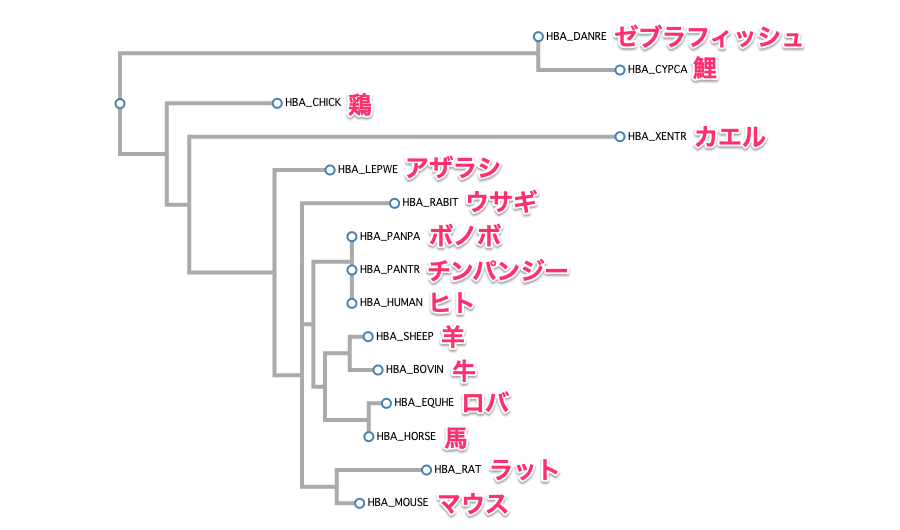
BIONJで系統樹を作成すると、次のようになります。
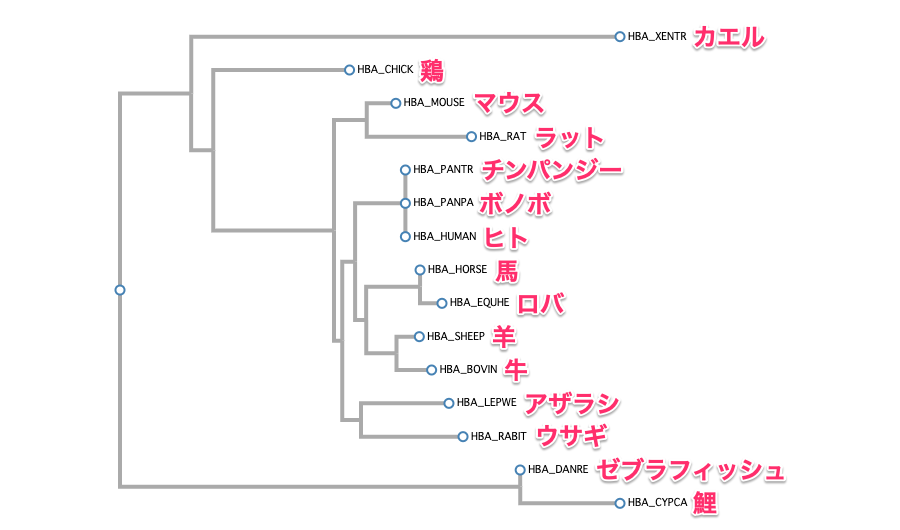
また、fasttreeで系統樹を作成すると、次のようになります。
演習 †
UniProtからEntry Nameが AMY1_ で始まるアミラーゼのアミノ酸配列をいくつか入手し、ClustalWを用いて多重配列アラインメントを行って系統樹を作成してみよう。
参考文献 †
- 樋口 千洋. 2011. Rによるバイオインフォマティクスデータ解析-Bioconductorを用いたゲノムスケールのデータマイニング, 第2版. 共立出版. https://amzn.to/4nz0g7v
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)