- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- バイオ・データ・マイニング/Rで相関分析する へ行く。
- 1 (2013-11-14 (木) 16:51:27)
- 2 (2013-11-14 (木) 17:05:58)
- 3 (2013-11-15 (金) 08:10:20)
- 4 (2013-11-19 (火) 10:59:00)
- 5 (2013-11-19 (火) 16:04:51)
- 6 (2013-11-21 (木) 08:56:55)
- 7 (2015-11-11 (水) 12:51:57)
- 8 (2016-11-16 (水) 14:24:21)
- 9 (2016-11-16 (水) 14:24:21)
- 10 (2016-11-16 (水) 14:24:21)
- 11 (2016-11-16 (水) 14:24:21)
- 12 (2019-10-04 (金) 17:52:06)
はじめに †
ここでは,Rを使って相関係数を求め,統計的有意性を検定する方法を説明します.
『Rによるバイオインフォマティクスデータ解析』の3.3節「基本統計関数」に少しだけ出てきます.
準備 †
Rのインストールについては,次のページを見てください.
ここでは,標準で使用できるirisデータセットを使います.
data(iris)
このデータセットは,アヤメの種類(Species)を花びらの長さ(Sepal.Length),幅(Lepal.Width),がくの長さ(Petal.Length),幅(Petal.Width)によって分類する問題です. 長さと幅は連続値,種類はsetosa, versicolor, virginicaのいずれかをとる離散値です.
このデータセットには,setosa, versicolor, virginicaという3種類のアヤメについて,それぞれ50個ずつ,合計150個のデータが含まれています. ランダムに10個のデータを選択して,見てみましょう.
iris[sort(sample(1:150,10)),]
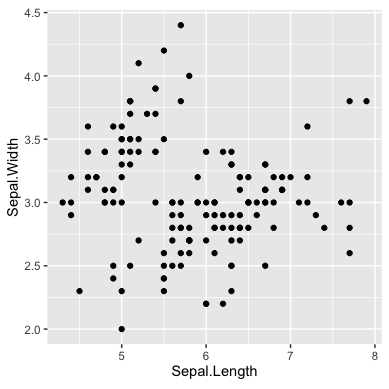
がく片(萼片)の長さと幅をプロットしてみます.
> iris[sort(sample(1:150,10)),] Sepal.Length Sepal.Width Petal.Length Petal.Width Species 4 4.6 3.1 1.5 0.2 setosa 22 5.1 3.7 1.5 0.4 setosa 65 5.6 2.9 3.6 1.3 versicolor 97 5.7 2.9 4.2 1.3 versicolor 100 5.7 2.8 4.1 1.3 versicolor 108 7.3 2.9 6.3 1.8 virginica 116 6.4 3.2 5.3 2.3 virginica 122 5.6 2.8 4.9 2.0 virginica 136 7.7 3.0 6.1 2.3 virginica 146 6.7 3.0 5.2 2.3 virginica
共分散 †
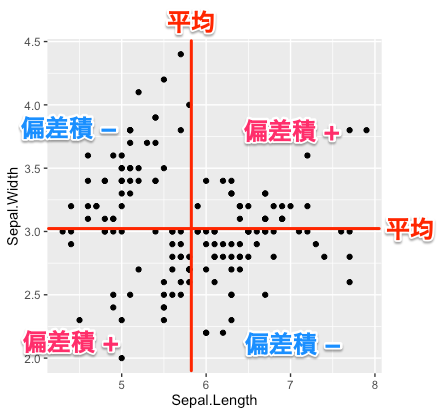
[math]X[/math] と [math]Y[/math] の共分散は,それぞれの平均値からのズレ(偏差)の積の平均です. \[ \mathrm{Cov}(X,Y) = \frac{\sum_{i=1}^{n} (x_i - \bar{X})(y_i - \bar{Y})}{n} \] ここで,[math]n[/math] はデータ数,[math]\bar{X}[/math], [math]\bar{Y}[/math] は,それぞれ,[math]X[/math], [math]Y[/math] の平均です.
偏差の符号が同じとき,つまり,どちらも平均より大きいかどちらも平均より小さいとき,共分散は大きくなり,偏差の符号が異なるとき,つまり,いずれかが平均より大きくもう一方が平均より小さいとき,共分散は小さくなります.
共分散を求めるには,cov関数を使います.
install.packages("ggplot2") library(ggplot2)
相関係数 †
[math]X[/math] と [math]Y[/math] の相関係数は,[math]X[/math] と [math]Y[/math] の共分散を [math]X[/math] の不偏標準偏差と [math]Y[/math] の不偏標準偏差の積で割ったものです. \[ \mathrm{Cor}(X, Y) = \frac{\mathrm{Cov}(X, Y)}{\mathrm{Var}(X) \mathrm{Var}(Y)} \]
不偏標準偏差は正の値ですから,共分散が正なら相関係数も正,共分散が負なら相関係数も負になります.
相関係数の値は-1から1までの値をとります. 相関係数が1に近いほど強い正の相関があり,相関係数が-1に近いほど強い負の相関があります. 相関係数が0に近いときは相関がありません.
相関係数を求めるには,cor関数を使います.
ggplot(data=iris, aes(x=Sepal.Length, y=Sepal.Width))+ geom_point()+ theme(aspect.ratio=1)
相関の検定 †
がく片(萼片)の長さと幅の相関係数は-0.12で,ほとんど相関がないことがわかりました.
本当に相関は無いのでしょうか. 相関係数の値がゼロに近かったのはこのサンプルだから偶然そうなったということは考えられないでしょうか.
そこで,「がく片(萼片)の長さと幅には相関がない」という仮説を立て,これが成り立つかどうかを調べます. これを無相関検定といいます. 詳しくは統計の教科書で勉強してください.
具体的には,p値を求め,有意水準と比べます. p値が有意水準より小さいとき,その水準において有意な相関があると考えます. そうでないとき,その水準において有意な相関はないと考えます. 有意水準には,5%や1%がよく用いられます.
p値を求めるには,cor.test関数を使います.
cov(iris$Sepal.Length, iris$Sepal.Width)
p値 (p-value) が0.15なので,1%有意水準でも,5%有意水準でも,相関があるとは言えません.
そこで,setosaのデータだけを取り出して,がく片(萼片)の長さと幅の相関を調べてみましょう.
> cov(iris$Sepal.Length, iris$Sepal.Width) [1] -0.042434
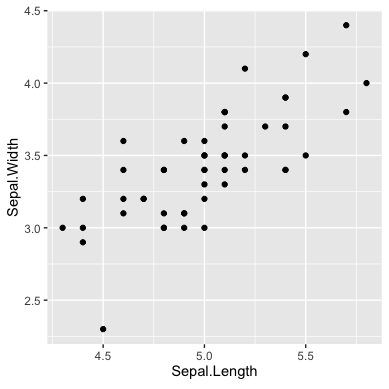
相関係数が0.74,p値は6.7e-10,つまり0.00000000067だったので,setosaのがく片(萼片)の長さと幅の間には1%水準で(0.0000001%水準でも)有意にかなり強い正の相関があると言えます.
サンプル数が少ないと,p値が大きくなる傾向にあります.
たとえば,setosaの1番目から5番目のデータだけを取り出して,がく片(萼片)の長さと幅の相関を調べてみます.
cor(iris$Sepal.Length, iris$Sepal.Width)
#ref(): File not found: "s1.png" at page "バイオ・データ・マイニング/Rで相関分析する"
すると,相関係数は0.68ですが,p値が0.21なので,有意な正の相関があるとは言えません.
また,21番目から25番目までのデータだけを取り出して,同じように相関を調べてみると,(setosaに含まれる50個のデータからは有意な正の相関が見られたのに)相関係数が-0.30と負になってしまいますが,p値が0.63ですから,これも有意な負の相関があるとは言えません.
> cor(iris$Sepal.Length, iris$Sepal.Width) [1] -0.1175698
#ref(): File not found: "s2.png" at page "バイオ・データ・マイニング/Rで相関分析する"
このように,サンプルの取り方によっては相関係数が大きく変わってしまいますので,p値を調べることによって,その相関が統計的に有意な相関であるかどうかを調べる必要があります.
参考文献 †
この本の3.3節「基本統計関数」に少しだけ出てきます.
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)