- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- バイオ・データ・マイニング/Rで主成分分析する へ行く。
このページはまだ書きかけです
はじめに †
準備 †
データ
> data(iris)
主成分分析 †
主成分分析は,多変量解析の一手法で,高次元の数値データに対して合成成分を作る手法です.
この合成成分のことを主成分といい,次のように表されます. \[ \begin{array}{c@{}c@{}c} z_1 &=& a_{1,1} x_{1} + a_{1,2} x_{2} + \dots + a_{1,p} x_{p} \\ z_2 &=& a_{2,1} x_{1} + a_{2,2} x_{2} + \dots + a_{2,p} x_{p} \\ \vdots & & \\ z_p &=& a_{p,1} x_{1} + a_{p,2} x_{2} + \dots + a_{p,p} x_{p} \end{array} \] ここで,[math]z_1[/math] を第1主成分,[math]z_2[/math] を第2主成分,つまり,[math]z_i[/math] を第 [math]i[/math] 主成分といいます.
次元数が [math]p[/math] のとき,主成分は [math]p[/math] 個できます. このうちの第1主成分からいくつかだけを使ってデータを表現できないかということを考えます.
例として,次のような2次元のデータを考えてみます*1.
2次元の場合,主成分(合成成分)は次のようになります. (図では変数が [math]x[/math], [math]y[/math] になっていますが,これを [math]x_1[/math], [math]x_2[/math]とします.) \[ \begin{array}{c@{}c@{}c} z_1 &=& a_{1,1} x_{1} + a_{1,2} x_{2} \\ z_2 &=& a_{2,1} x_{1} + a_{2,2} x_{2} \end{array} \]
このデータは2変数で表現されていますが,これを第1主成分だけで表現するとしたら,どのように第1主成分を選ぶ,つまり,どのように第1主成分の係数 [math]a_{1,1}[/math], [math]a_{1,2}[/math] を選ぶのがいいのかを考えます.
合成成分である第1主成分だけでデータを表現するということは,データを1次元で表現するということですから,データから第1主成分を表す直線に垂線を下ろし,その値でデータを表現するということになります.
下の図のように,データから第1主成分である直線に垂線を下ろしたときに同じような場所になってしまう,つまり,分散が小さいのはよくありません.
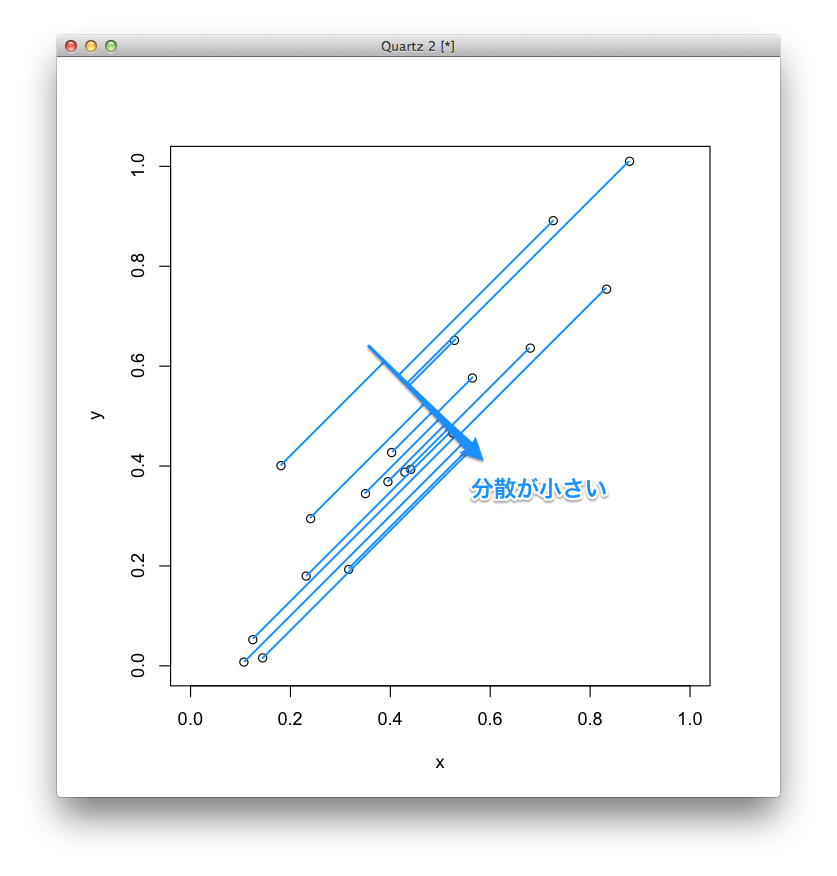
逆に,下の図のように,データから第1主成分である直線に垂線を下ろしたときにバラツキが大きくなる,つまり,分散が大きくなるほうが,データを良く表していると考えます.
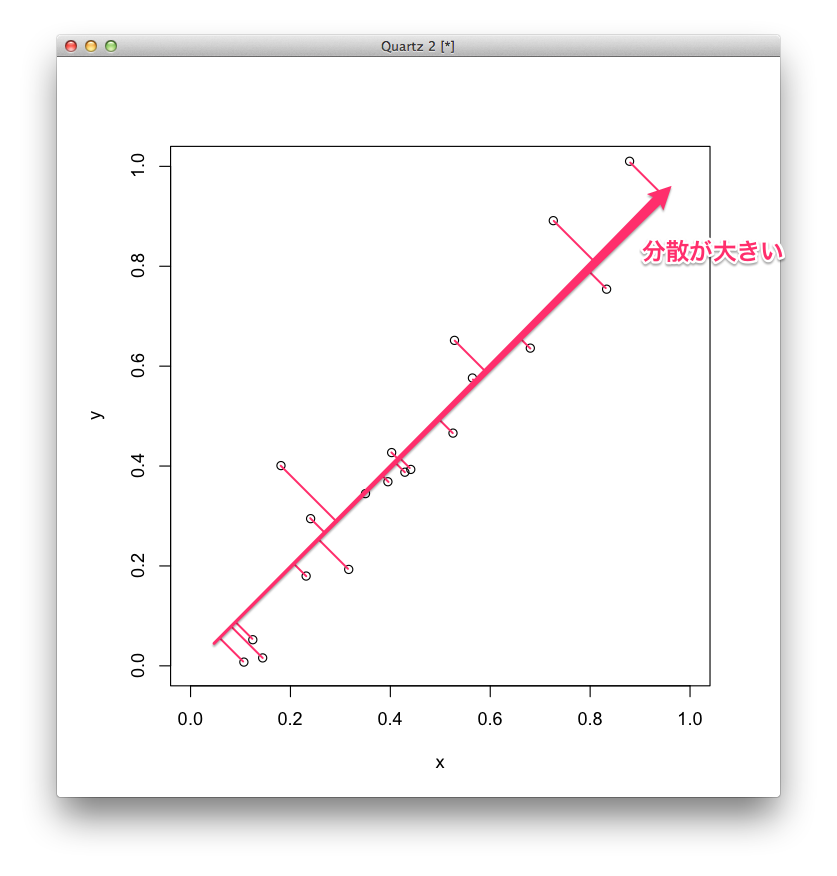
そこで,垂線を下ろしたときに分散が最も大きくなる直線を第1主成分とします.
第2主成分は,第1主成分と直行する直線の中で,データから垂線を下ろしたときに分散が最も大きくなる直線とし,以後同様に,第 [math]i+1[/math] 成分は第 [math]i[/math] 主成分と直行する直線の中で,データから垂線を下ろしたときに分散が最も大きくなる直線とします. 最後の主成分である第 [math]p[/math] 主成分は,選ぶことができずに決まります.
今回の例は2次元なので,第2主成分は第1主成分に直行する直線に決まってしまい,下の図のようになります.
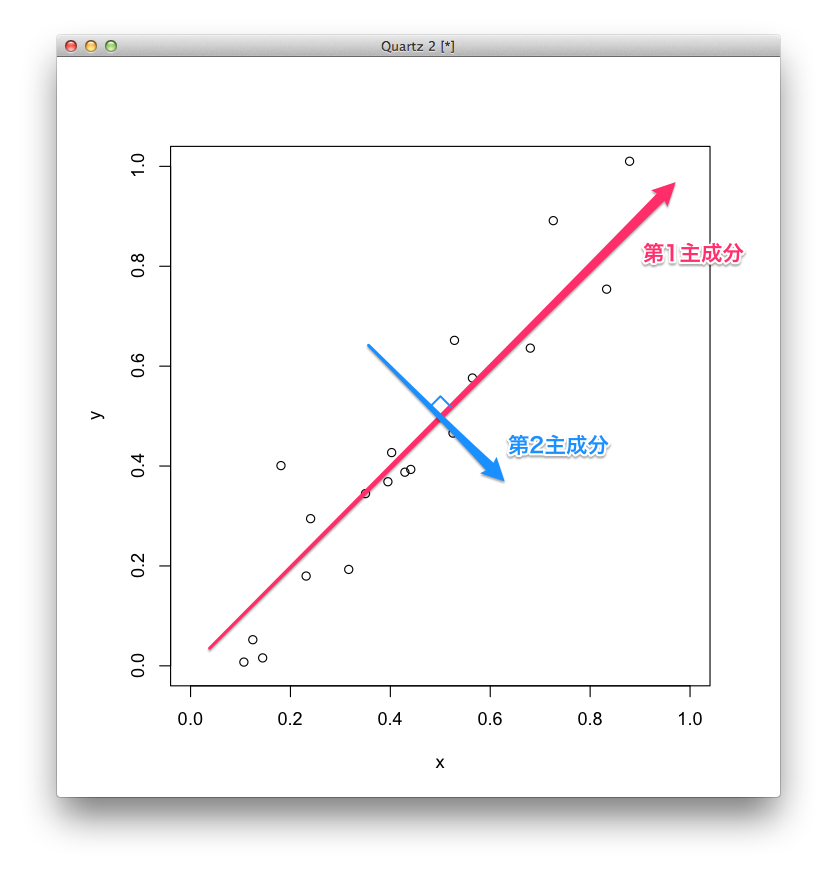
主成分を求めるには,まず各変数を平均0,標準偏差1に正規化し,分散共分散行列の固有値と固有ベクトルを求めます. 分散共分散行列は [math]p \times p[/math] 正方行列なので,固有値は [math]p[/math] 個求まり,大きい順に [math]\lambda_1[/math], [math]\lambda_2[/math], [math]\dots[/math], [math]\lambda_p[/math] とすると,[math]i[/math] 番目の固有値 [math]\lambda_i[/math] が第 [math]i[/math] 主成分の分散になります. 固有ベクトルは,主成分における正規化されたデータに対する係数を表します.
ある固有値を固有値全部の合計で割った値を主成分寄与率といいます. つまり,第 [math]i[/math] 主成分の寄与率は以下の式で表されます. \[ \frac{\lambda_i}{\sum_{j=1}^{p} \lambda_{j}} \] 固有値は分散を表していますから,寄与率は主成分全体の分散のうちその主成分の分散の割合を表しています.
また,主成分全体の分散のうち第1主成分から第 [math]i[/math] 主成分までの分散の和の割合を累積寄与率といい,以下の式で表されます. \[ \frac{\sum_{j=1}^{i} \lambda_j}{\sum_{j=1}^{p} \lambda_{j}} \] この累積寄与率が0.8あればいいとか,0.9あればいいというように決めて,どこまでの主成分を採用するかを決めます.
上の例では,第1主成分の分散は約1.96,第2主成分の分散は0.04です. したがって,第1主成分の寄与率は約0.98,第2主成分の寄与率は0.02です. また,累積寄与率は,第1主成分までで約0.98,第2主成分までで1となります.
ここで,累積寄与率が0.9まででいいとするなら,第2主成分は採用せずに第1主成分だけでデータを表現してもいいことになります. 当然のことですが,情報量は落ちるので注意が必要です.
主成分における正規化されたデータに対する係数を,主成分負荷量といいます. 主成分負荷量は,固有ベクトルに対応する主成分の標準偏差を掛けて求めます.
この図の例では,第1主成分の固有ベクトルが約 [math]\left(\begin{array}{r}0.707 \\ 0.707 \end{array} \right)[/math] で標準偏差が約1.4,第2主成分の固有ベクトルが約 [math]\left( \begin{array}{r} -0.707 \\ 0.707 \end{array}\right)[/math] で標準偏差が約0.2ですから,第1主成分に対する主成分負荷量は [math]x_1[/math] も [math]x_2[/math] も約0.990,第2主成分に対する [math]x_1[/math] の主成分負荷量は約-0.143,[math]x_2[/math] の主成分負荷量は0.143になります.
データを正規化して,第 [math]i[/math] 主成分に垂線を下ろしたところの値を第 [math]i[/math] 主成分の主成分スコアといいます.
図の一番左下のデータは,約 (0.107, 0.008) です. [math]x_1[/math] の平均は約0.46,標準偏差は約0.27で,[math]x_2[/math] の平均は約0.47,標準偏差は0.33ですから,これを正規化すると約 (-1.29, -1.38) になります. ここから第1主成分と第2主成分に垂線を下ろしたところの値が,それぞれの主成分スコアで約(0.08, -0.07)となります.
主成分分析を実行する †
主成分分析を実行
> iris[sort(sample(1:150,10)),] Sepal.Length Sepal.Width Petal.Length Petal.Width Species 4 4.6 3.1 1.5 0.2 setosa 22 5.1 3.7 1.5 0.4 setosa 65 5.6 2.9 3.6 1.3 versicolor 97 5.7 2.9 4.2 1.3 versicolor 100 5.7 2.8 4.1 1.3 versicolor 108 7.3 2.9 6.3 1.8 virginica 116 6.4 3.2 5.3 2.3 virginica 122 5.6 2.8 4.9 2.0 virginica 136 7.7 3.0 6.1 2.3 virginica 146 6.7 3.0 5.2 2.3 virginica
主成分分析の結果
> model <- princomp(~Sepal.Length+Sepal.Width+Petal.Length+Petal.Width, data=iris, cor=T)
固有ベクトル
> model Call: princomp(formula = ~Sepal.Length + Sepal.Width + Petal.Length + Petal.Width, data = iris, cor = T) Standard deviations: Comp.1 Comp.2 Comp.3 Comp.4 1.7083611 0.9560494 0.3830886 0.1439265 4 variables and 150 observations. > summary(model) Importance of components: Comp.1 Comp.2 Comp.3 Comp.4 Standard deviation 1.7083611 0.9560494 0.38308860 0.143926497 Proportion of Variance 0.7296245 0.2285076 0.03668922 0.005178709 Cumulative Proportion 0.7296245 0.9581321 0.99482129 1.000000000
固有値
> unclass(model$loadings) Comp.1 Comp.2 Comp.3 Comp.4 Sepal.Length 0.5210659 -0.37741762 0.7195664 0.2612863 Sepal.Width -0.2693474 -0.92329566 -0.2443818 -0.1235096 Petal.Length 0.5804131 -0.02449161 -0.1421264 -0.8014492 Petal.Width 0.5648565 -0.06694199 -0.6342727 0.5235971
因子寄与率(主成分寄与率)
> model$sd^2 Comp.1 Comp.2 Comp.3 Comp.4 2.91849782 0.91403047 0.14675688 0.02071484
因子負荷量(主成分負荷量)
> model$scores Comp.1 Comp.2 Comp.3 Comp.4 1 -2.26470281 -0.480026597 0.127706022 0.024168204 2 -2.08096115 0.674133557 0.234608854 0.103006775 3 -2.36422905 0.341908024 -0.044201485 0.028377053 4 -2.29938422 0.597394508 -0.091290106 -0.065955560 5 -2.38984217 -0.646835383 -0.015738196 -0.035922813 ...
tは行列を転置する関数です.
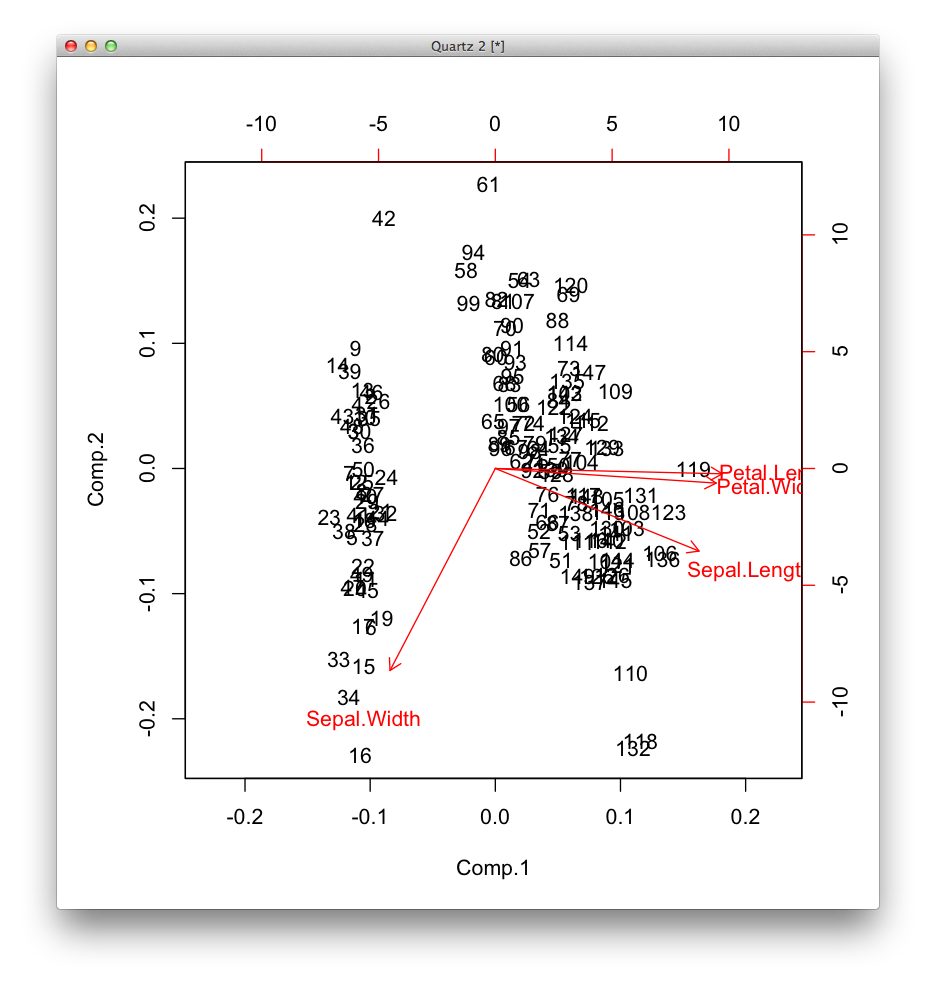
主成分分析の結果をグラフに表示する †
> biplot(model)
主成分スコアを求める †
データの行列と固有ベクトルの内積を求める
df <- data.frame(model$scores) df <- transform(df, Species=iris$Species) library(ggplot2) ggplot(data=df, aes(x=Comp.1, y=Comp.2, color=Species, shape=Species)) + geom_point() + theme(aspect.ratio=1)
%*% は行列の積.
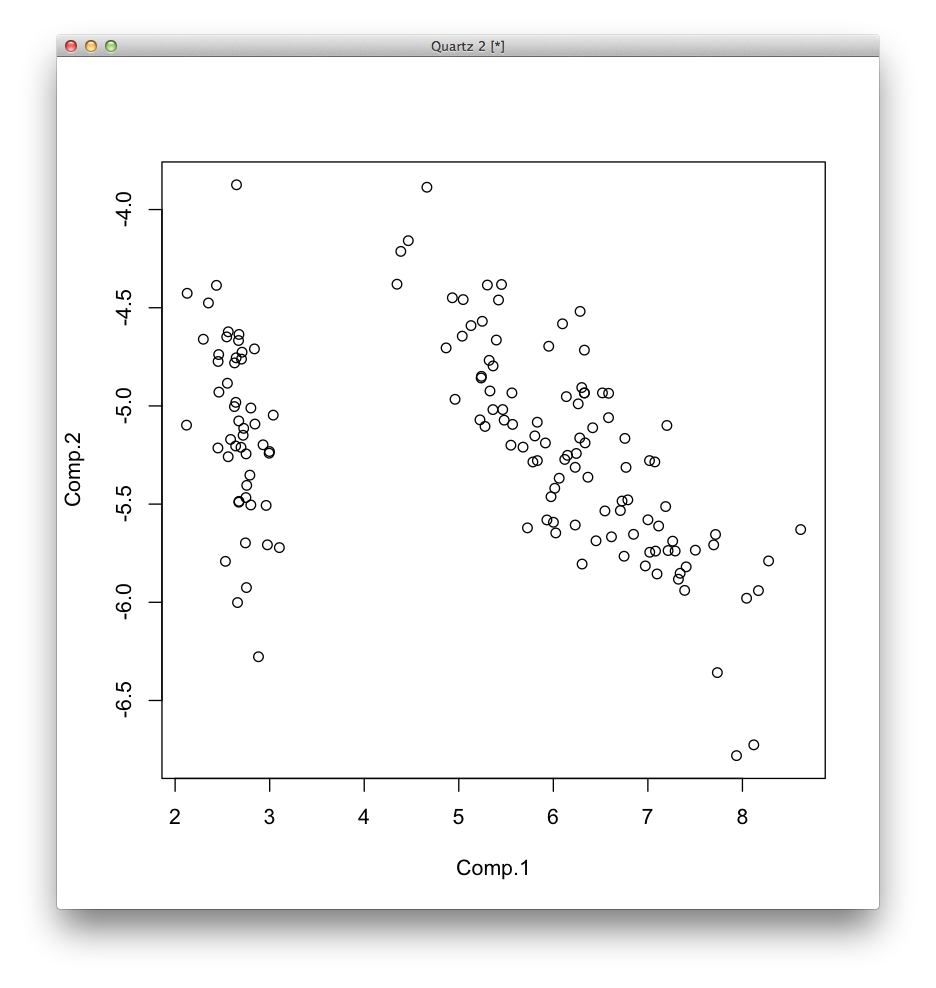
これをグラフにプロットする.
> plot(data.matrix(iris[,c(1,2,3,4)]) %*% unclass(loadings(iris.pc)[,c(1,2)]))
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)