- バックアップ一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- バイオ・データ・マイニング/Rで非線形回帰分析する へ行く。
- 1 (2015-02-01 (日) 11:36:18)
- 2 (2015-02-01 (日) 12:22:22)
このページは自分用のメモで,まだ書きかけです.
はじめに †
ここでは,Rを使って非線形回帰分析をします.
目的関数が線形の場合,最小二乗法を用いて残差 (RSS) が最小となるパラメーターを解析的に求めることができます. これから回帰分析をはじめる場合は,先に線形回帰分析をやってみましょう.
- Rで回帰分析する - とうごろうぃき
目的関数が非線形の場合,最小二乗法で解析的に解くことができません. そこで,ニュートン法や最急降下法のような方法を使って,残差 (RSS) が最小となるパラメーターを探索します. ここでは,Levenberg-Marquardt法を用います.
Levenberg-Marquardt法は,minpack.lmパッケージに含まれています.
非線形回帰 †
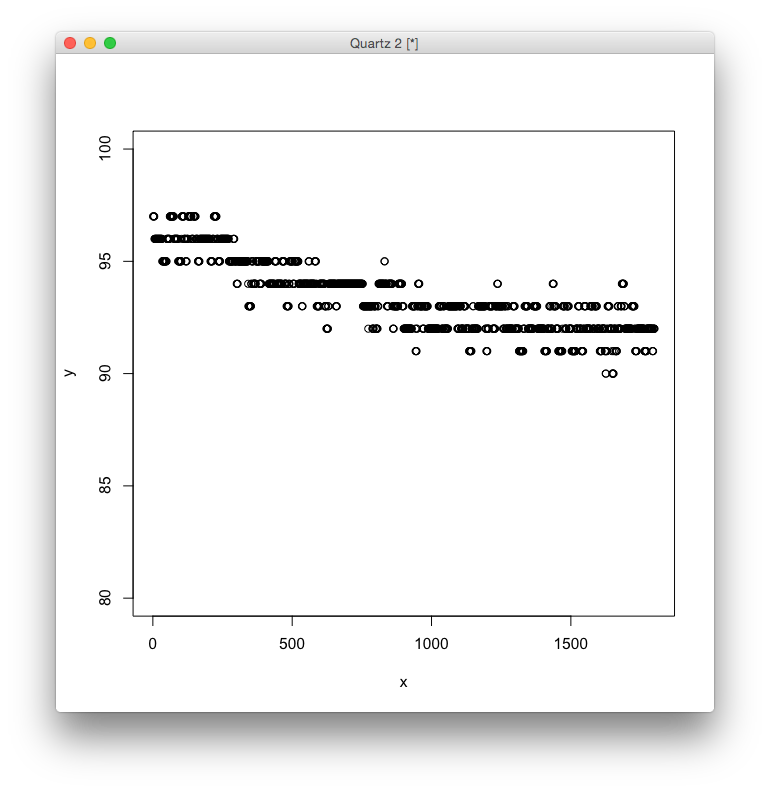
ここでは,次のようなデータに対して非線形回帰を行います.
> data <- read.csv("data.csv") > head(data) x y 1 1 97 2 2 97 3 3 97 4 4 97 5 5 97 6 6 96 > plot(x, y, ylim=c(80,100))
この値はゆるやかに単調に減少し,収束するように見えるので,次のような式で表される曲線に当てはめてみます. \[ y = a e^{bx} + c \]
ちなみに,ゼロに収束する場合は [math]c = 0[/math] となり,両辺の対数を取ることで線形に変換して線形回帰で解くことができます.
まず,当てはめたい関数を定義します.
> getPred <- function(parS, xx) parS$a * exp(xx * parS$b) + parS$c
次に,残差 (RSS) を求める関数を定義します.
> residFun <- function(p, observed, xx) observed - getPred(p,xx)
それから,探索するパラメーターの初期値を定義します.
> parStart <- list(a=10,b=-.001, c=90)
最後に,nls.lm関数を用いてパラメーターを探索します.
> nls.out <- nls.lm(par=parStart, fn=residFun, observed=data$y, xx=data$x, + control=nls.lm.control(nprint=1)) It. 0, RSS = 5260.3, Par. = 10 -0.001 90 It. 1, RSS = 968.102, Par. = 5.45869 -0.00112816 91.2747 It. 2, RSS = 900.171, Par. = 5.56857 -0.00120959 91.1581 It. 3, RSS = 900.134, Par. = 5.58886 -0.00119717 91.1308 It. 4, RSS = 900.134, Par. = 5.58758 -0.00119872 91.1333 It. 5, RSS = 900.134, Par. = 5.58777 -0.00119853 91.133 > summary(nls.out) Parameters: Estimate Std. Error t value Pr(>|t|) a 5.588e+00 8.899e-02 62.79 <2e-16 *** b -1.199e-03 5.788e-05 -20.71 <2e-16 *** c 9.113e+01 1.080e-01 843.73 <2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Residual standard error: 0.7077 on 1797 degrees of freedom Number of iterations to termination: 5 Reason for termination: Relative error in the sum of squares is at most `ftol'.
探索によって見つかった最良のパラメーターは出力されたオブジェクトのpar変数に入っています.
> nls.out$par $a [1] 5.587771 $b [1] -0.001198529 $c [1] 91.13298
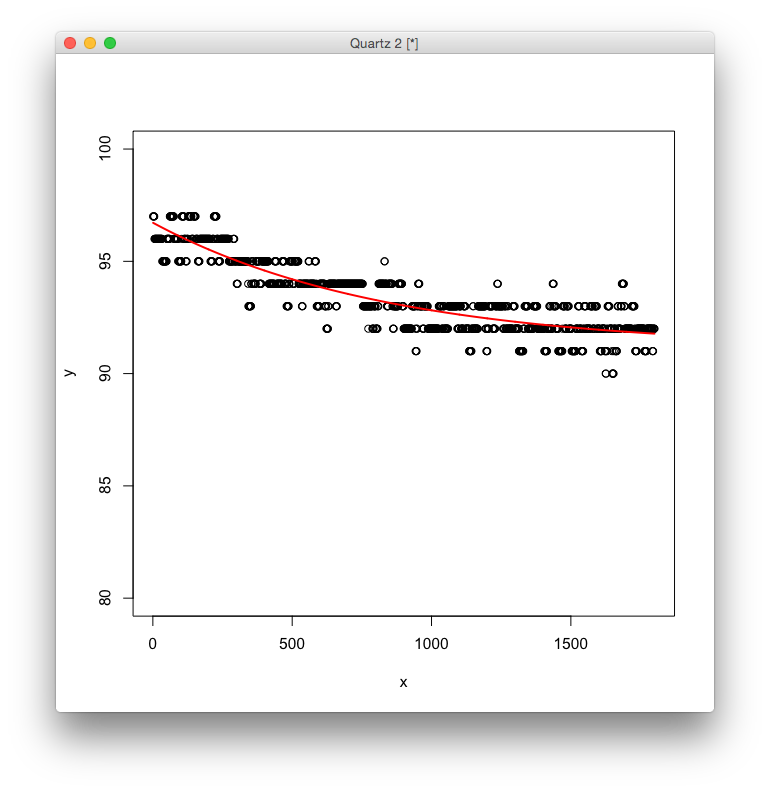
求めた曲線をデータに重ねてみます.
> lines(data$x, getPred(as.list(coef(nls.out)), data$x), col=2, lwd=2)
![[PukiWiki] [PukiWiki]](https://xn--p8ja5bwe1i.jp:443/wiki/image/banner.png)